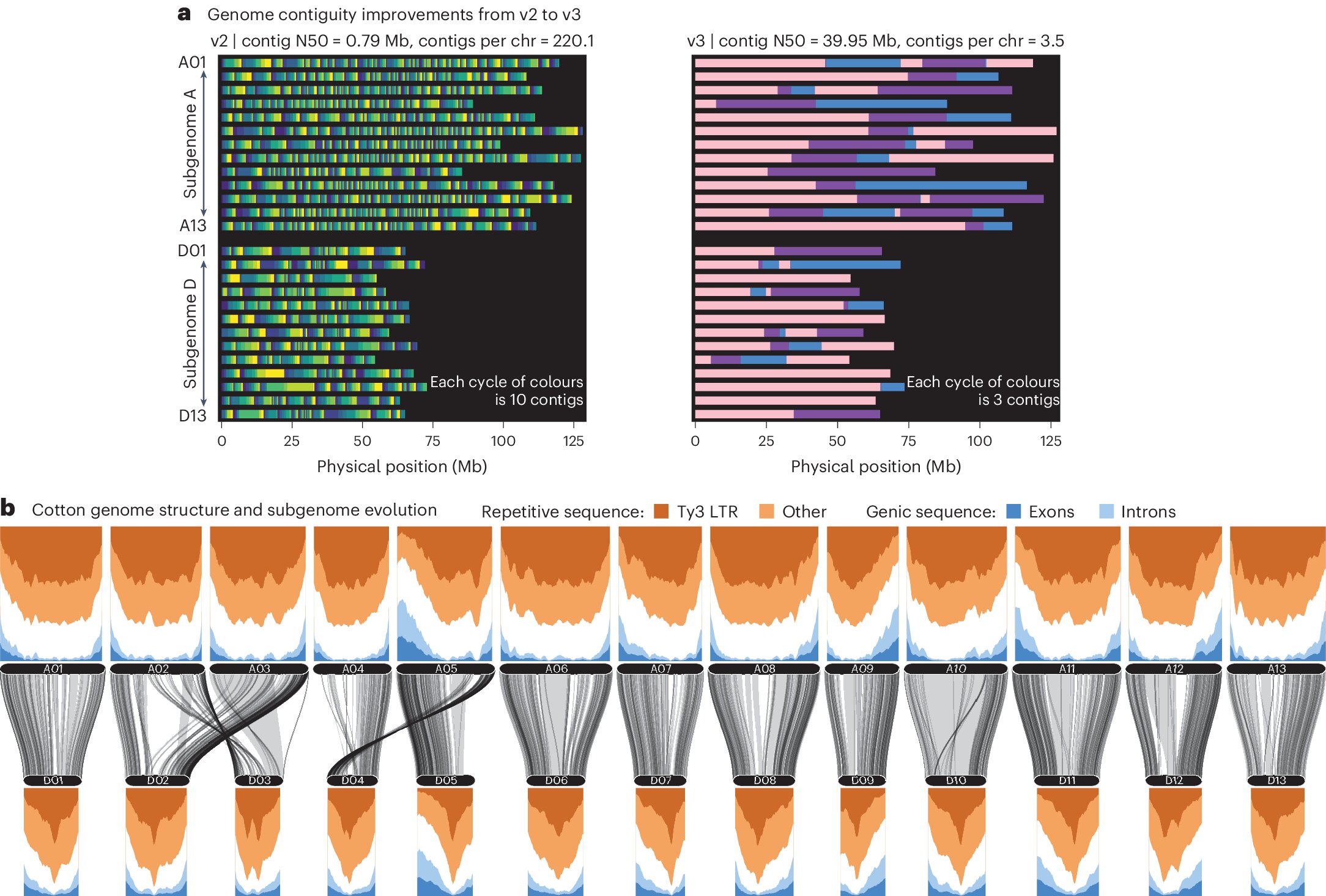
Struktur und Kontiguität des Baumwoll-TM-1-Referenzgenoms. Die genomischen Referenzsequenzen v2 und v3 wurden einer Contig-Positionskartierung durch GENESPACE unterzogen. a, Die Contigs in jedem Genom (v2, links; v3, rechts) als kontinuierlicher Block einer einzelnen Farbe. Angesichts der erheblichen Unterschiede in der Nachbarschaft wurde für Version 2 eine kontinuierliche Gelb-Blau-Palette mit zehn Farben ausgewählt, während für Version 3 eine diskrete Abfolge von drei Farben (Rosa, Lila, Blau) verwendet wurde. b, Der Unterschied in der Genomarchitektur zwischen den Subgenomen A (oben) und D (unten) der tetraploiden Baumwolle TM-1 v3. Wiederholungen und Gendichte wurden hierarchisch abgeleitet, wobei Genome in Exons, Ty3-Wiederholungen, andere Wiederholungen (von RepeatMasker), Introns und andere (leer) klassifiziert wurden. Es werden Schiebefenster (5 MB breit, 1 MB-Schritte) gezeichnet. Zwischen den beiden Subgenomen werden zerlegte Blöcke von Minimap2-Alignments angezeigt. Kredit: Natürliche Pflanzen (2024). DOI: 10.1038/s41477-024-01713-z
Wir leben in einer sich ständig verändernden und wachsenden Welt. Der Klimawandel, das Auftreten von Schädlingen und andere Umweltstressoren setzen die Nutzpflanzen, die die Welt ernähren und mit Energie versorgen, unter Druck. Da wir bestrebt sind, die wachsende Nachfrage nach hochwertigen, nachhaltigen Nahrungsmitteln und Faserpflanzen zu decken, erweist sich die Genomik als wirksames Instrument im Kampf. Durch das Verständnis der genetischen Codes von Pflanzen können Forscher und Züchter Nutzpflanzen mit höheren Erträgen, verbesserter Resistenz gegen Schädlinge und Krankheiten und größerer Anpassungsfähigkeit an Umweltherausforderungen entwickeln.
Genombasierte Züchtung kommt vor allem Nutzpflanzen zugute, die bereits über hochwertige genomische Ressourcen verfügen, etwa Reis und Weizen. Allerdings müssen Pflanzen mit weniger ausgereiften genomischen Ressourcen weiterhin auf traditionelle Züchtungsmethoden zurückgreifen, die manchmal unter einem Mangel an genomischer Vielfalt innerhalb der Zuchtpopulationen leiden.
Baumwolle, eine weltweit lebenswichtige Nutzpflanze, verfügt nicht über robuste genomische Ressourcen. Die Baumwollindustrie ist ein großes Geschäft mit einem globalen wirtschaftlichen Einfluss von 600 Milliarden US-Dollar und bietet Arbeitsplätze für mehr als 250 Millionen Menschen. Für eine erfolgreiche Baumwollproduktion sind Baumwollsorten mit wünschenswerten Eigenschaften wie hohem Ertrag, guter Faserqualität, Schädlings- und Krankheitsresistenz und Trockenheitstoleranz erforderlich.
„Baumwollzüchter haben im Laufe der Jahre mit traditionellen Züchtungsmethoden die Faserausbeute und -qualität verbessert“, sagt Jeremy Schmutz, Co-Direktor des HudsonAlpha Genome Sequencing Center, der sich seit über einem Jahrzehnt mit der Baumwollgenomik beschäftigt. „Es könnte für sie schwierig sein, zusätzliche Verbesserungen zu erzielen, da es bei moderner domestizierter Baumwolle keine genetische Variation gibt. Die Entwicklung neuer genomischer Werkzeuge für die Industrie wird dazu beitragen, die Verbesserungen bei Baumwolle auf die nächste Stufe zu heben.“
Wissenschaftler am Genome Sequencing Center (GSC) des HudsonAlpha Institute of Biotechnology und andere Mitarbeiter haben sich zum Ziel gesetzt, hochwertige Genomsequenzen für drei wichtige Baumwollsorten zu erstellen und so den Baumwollzüchtern die benötigten genomischen Ressourcen bereitzustellen. Die Ergebnisse wurden kürzlich in veröffentlicht Natürliche Pflanzen.
„Die Baumwollforschung hat sich stark auf ein Referenzgenom, ‚TM1‘, gestützt, eine Baumwollsorte, die in Zuchtprogrammen nicht mehr weit verbreitet ist“, sagt Erstautor Avinash Sreedasyam, Ph.D. „Damit die molekulare Züchtung der Baumwollindustrie zugute kommt, müssen viele unterschiedliche Genome vorhanden sein, um die Vielfalt der Baumwollsorten darzustellen. Diese Studie generierte hochwertige Referenzgenome für drei moderne Hochland-Baumwollsorten und aktualisierte die Genetik der „TM-1“-Baumwolle. Standardreferenz.“
Die erste Analyse der neuen Referenzgenome lieferte wichtige Informationen zur Faserqualität. Die hochpräzisen und umfassenden Genomassemblierungen wurden verwendet, um das genetische Material der Pima-Baumwolle (bekannt für die überlegene Qualität ihrer Fasern) innerhalb moderner Baumwollsorten zu identifizieren. Kleine Abschnitte jedes Genoms wurden sowohl mit dem Pima-Genom als auch mit dem Referenz-Baumwollgenom verglichen.
Segmente, die eher mit Pima als mit Referenzbaumwolle übereinstimmten, wurden als potenzielle Introgressionen eingestuft, was darauf hindeutet, dass Pima-DNA in die genetische Ausstattung moderner Baumwolle eingebaut wurde. Die Kenntnis dieser Pima-Introgressionen wird Züchtern dabei helfen, in ihren Zuchtprogrammen effektiv Nachkommen mit diesen faserqualitätsbezogenen genetischen Markern auszuwählen.
„Die Nutzung relativ kostengünstiger Low-Pass-Sequenzierung neben diesen Genomen ermöglicht es Züchtern, ihre Nachkommen schnell auszuwählen“, sagt Sreedasyam. „Dies wird nicht nur Zeit sparen, sondern auch die Kosten senken, die mit der herkömmlichen Faserphänotypisierung verbunden sind, einem mühsamen Prozess, der typischerweise Hunderte oder sogar Tausende von Proben pro Screening-Durchlauf erfordert.“
Diese Ergebnisse unterstreichen die Bedeutung der Verwendung detaillierter Genomassemblierungen zur Entdeckung genetischer Variationen, die Baumwollzuchtprogramme verbessern können. Je häufiger diese neuen hochwertigen Genome für vergleichende Studien verwendet werden, desto mehr Informationen über die wirtschaftlich wichtigen Merkmale der Baumwolle werden gewonnen. Die in dieser Studie beschriebenen genomischen Ressourcen stellen eine wertvolle Ergänzung des Werkzeugkastens für die Baumwollzüchtung dar und werden in den kommenden Jahren von Nutzen sein.
Zu den Mitarbeitern dieses Projekts gehören Don C. Jones, Cotton Incorporated, North Carolina; Peng W. Chee, University of Georgia, Tifton, Georgia; Warwick N. Stiller, CSIRO, Cotton Research Unit, Australien; und Fred Bourland, University of Arkansas, Keiser, AR.
Mehr Informationen:
Avinash Sreedasyam et al.: Genomische Ressourcen dreier moderner Baumwolllinien leiten zukünftige Zuchtbemühungen, Natürliche Pflanzen (2024). DOI: 10.1038/s41477-024-01713-z
Bereitgestellt vom HudsonAlpha Institute of Biotechnology
Zitat: Neue genomische Werkzeuge für drei moderne Baumwollsorten könnten zukünftige Züchtungsbemühungen leiten (30. Mai 2024), abgerufen am 30. Mai 2024 von https://phys.org/news/2024-05-genomic-tools-modern-cotton -varieties. html
Dieses Dokument unterliegt dem Urheberrecht. Abgesehen von der angemessenen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.