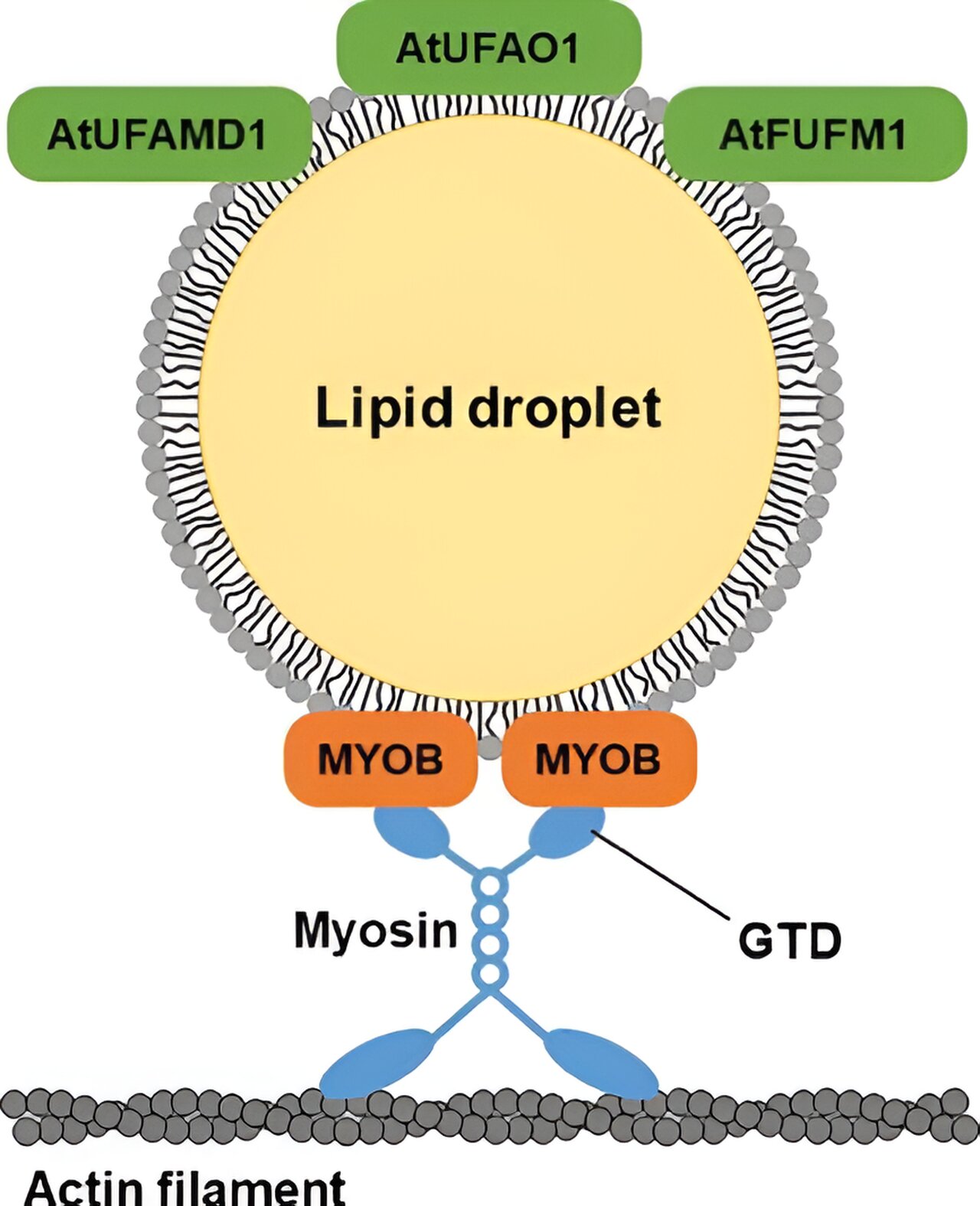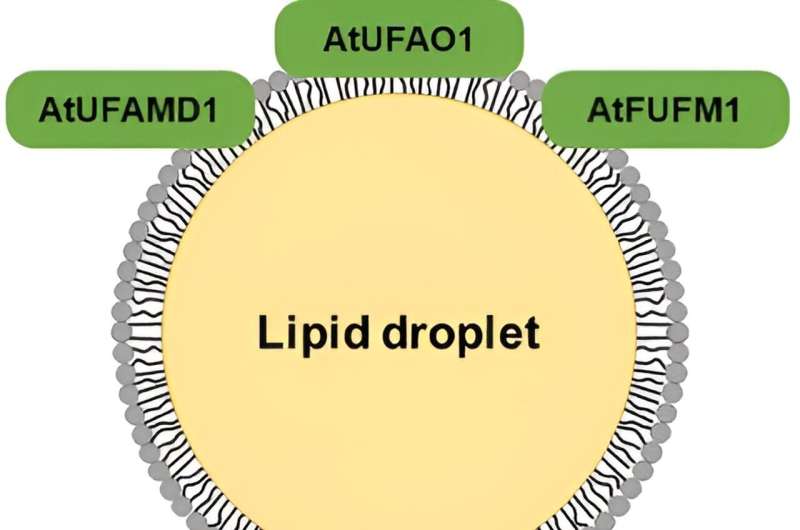
Ein hypothetisches Modell LD-lokalisierter Proteine. LD-lokalisierte MYOBs für die Bewegung von LDs und LD-lokalisierte Enzyme für die Biosynthese furanhaltiger Fettsäuren. Kredit: Grenzen der Pflanzenwissenschaft (2024). DOI: 10.3389/fpls.2024.1331479
Lipide sind Biomoleküle, die für das reibungslose Funktionieren lebender Zellen unerlässlich sind. Sie kommen in Zellmembranen vor und sind auch ein integraler Bestandteil der Zellsignalwege. Pflanzenzellen verfügen über subzelluläre Strukturen oder Organellen, sogenannte Lipidtröpfchen (LDs), in Blättern und Samen, die überschüssige Lipide (Fette) speichern.
Aktuelle Studien haben gezeigt, dass LDs auch einzigartige Pflanzenproteine lokalisieren, die wesentliche molekulare Funktionen erfüllen. Beispielsweise lokalisieren LDs in Samen Pflanzenproteine, sogenannte Oleosine, die den Samen dabei helfen, Frosttemperaturen zu widerstehen und richtig zu keimen.
Angesichts der zunehmenden Belege dafür, dass LDs nicht nur Organellen zur Kohlenstoffspeicherung sind, sondern auch wesentliche molekulare Funktionen erfüllen können, versuchte eine Gruppe japanischer Forscher, die Rolle relativ unerforschter Blatt-LDs zu verstehen.
Associate Professor Takashi L. Shimada, ein LD-Experte der Universität Chiba, erklärt die Motivation hinter ihrer Forschung. „Wir haben ein begrenztes Verständnis der Funktionen von Blatt-LDs: Warum reichern Pflanzenblätter LDs an? Wir glauben, dass die Beantwortung dieser grundlegenden Frage auch zur Technologie der Lipidproduktion beitragen würde.“
In einer neuen Studie veröffentlicht in Grenzen der PflanzenwissenschaftDr. Shimada und seine Forschungsgruppe zeigten, dass Blatt-LDs Myosin-bindende Proteine lokalisieren, die für intrazelluläre Bewegungen wichtig sind, sowie Enzyme, die die Furanfettsäure-Biosynthese – eine Art Fettproduktion in lebenden Zellen – katalysieren, was das Potenzial und die bisher unbekannten molekularen Funktionen von LDs hervorhebt.
Diese Forschung wurde von Dr. Shimada geleitet, der mit der Graduate School of Horticulture, dem Plant Molecular Science Center und dem Research Center for Space Agriculture and Horticulture an der Universität Chiba, Japan, verbunden ist.
Zu seiner Forschungsgruppe gehörten auch Herr Yuto Omata von der Fakultät für Gartenbau der Universität Chiba, der Erstautor dieser Studie, sowie Dr. Emi Mishiro-Sato, Dozent am World Premier International Research Center Initiative-Institute of Transformative Bio-Molecules, Universität Nagoya, sowie Dr. Ikuko Hara-Nishimura und Dr. Haruko Ueda von der Fakultät für Naturwissenschaften und Technik der Konan-Universität in Japan.
Die Forscher verwendeten ein Arabidopsis-Pflanzenmodell, das mutiert war, um LDs in Blättern anzusammeln, den sogenannten Arabidopsis thaliana-Mutanten-High-Sterol-Ester 1. Dr. Shimada fügt hinzu: „Dieses Arabidopsis-Mutanten-Pflanzenmodell dient auch als wertvolle Ressource für weitere Untersuchungen.“ Blatt-LD-Proteom. »
Darüber hinaus isolierten die Forscher diese LDs aus Blättern und verwendeten Massenspektrometrie, um assoziierte Proteine zu identifizieren. Sie fanden 3.206 Kandidatenproteine. Sie konzentrierten sich auf 31 Kandidatenproteine, um das damit verbundene Verhalten in Blatt-LDs zu testen. Sie brachten an diesen Proteinen eine Fluoreszenzmarkierung an, um mikroskopische Beobachtungen zu erleichtern.
Fluoreszenzmikroskopie zeigte das Vorhandensein von MYOSIN BINDING PROTEIN14 (MYOB14) und zwei nicht charakterisierten Proteinen in den LDs der untersuchten Blätter. Durch weitere Untersuchungen konnten die in LD-Blättern lokalisierten Mitglieder der MYOB-Familie eingegrenzt werden: MYOB1, MYOB2, MYOB3 und MYOB5.
Die Forscher identifizierten die beiden nicht charakterisierten Proteine auch als Enzyme für die Furanfettsäure-Biosynthese, was auf einen möglichen Zusammenhang zwischen Blatt-LDs und der Art der Fettsäureproduktion schließen lässt.
Trotz der in dieser Studie erzielten Fortschritte warnt Dr. Shimada: „Wir müssen zusätzliche LD-Proteine identifizieren, um die molekularen und physiologischen Funktionen von Blatt-LDs aufzuklären.“ »
Nichtsdestotrotz ebnet die Studie den Weg für zukünftige Forschungen zu Blatt-LDs – einer relativ unerschlossenen Pflanzenressource – die dank der Bemühungen von Dr. Shimada und seinem Team die Lipidproduktion und damit verbundene Industrien revolutionieren könnte.
Mehr Informationen:
Yuto Omata et al., Lipidtröpfchen aus Blättern von Arabidopsis thaliana enthalten Myosin-bindende Proteine und Enzyme, die mit der Biosynthese furanhaltiger Fettsäuren verbunden sind. Grenzen der Pflanzenwissenschaft (2024). DOI: 10.3389/fpls.2024.1331479
Zur Verfügung gestellt von der Universität Chiba
Zitat: Enthüllung molekularer Funktionen von Lipidtröpfchenproteinen in Blättern von Arabidopsis thaliana (23. April 2024), abgerufen am 23. April 2024 von https://phys.org/news/2024-04-unveiling-molecular-functions-lipid-droplet.html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.