Kredit: Naturprotokolle (2024). DOI: 10.1038/s41596-024-01046-3
Ein Forschungsteam unter der Leitung von Wissenschaftlern der University of California, Riverside, hat einen rechnerischen Arbeitsablauf zur Analyse großer Datensätze im Bereich der Metabolomik entwickelt, der Untersuchung kleiner Moleküle, die in Zellen, Bioflüssigkeiten, Geweben und ganzen Ökosystemen vorkommen.
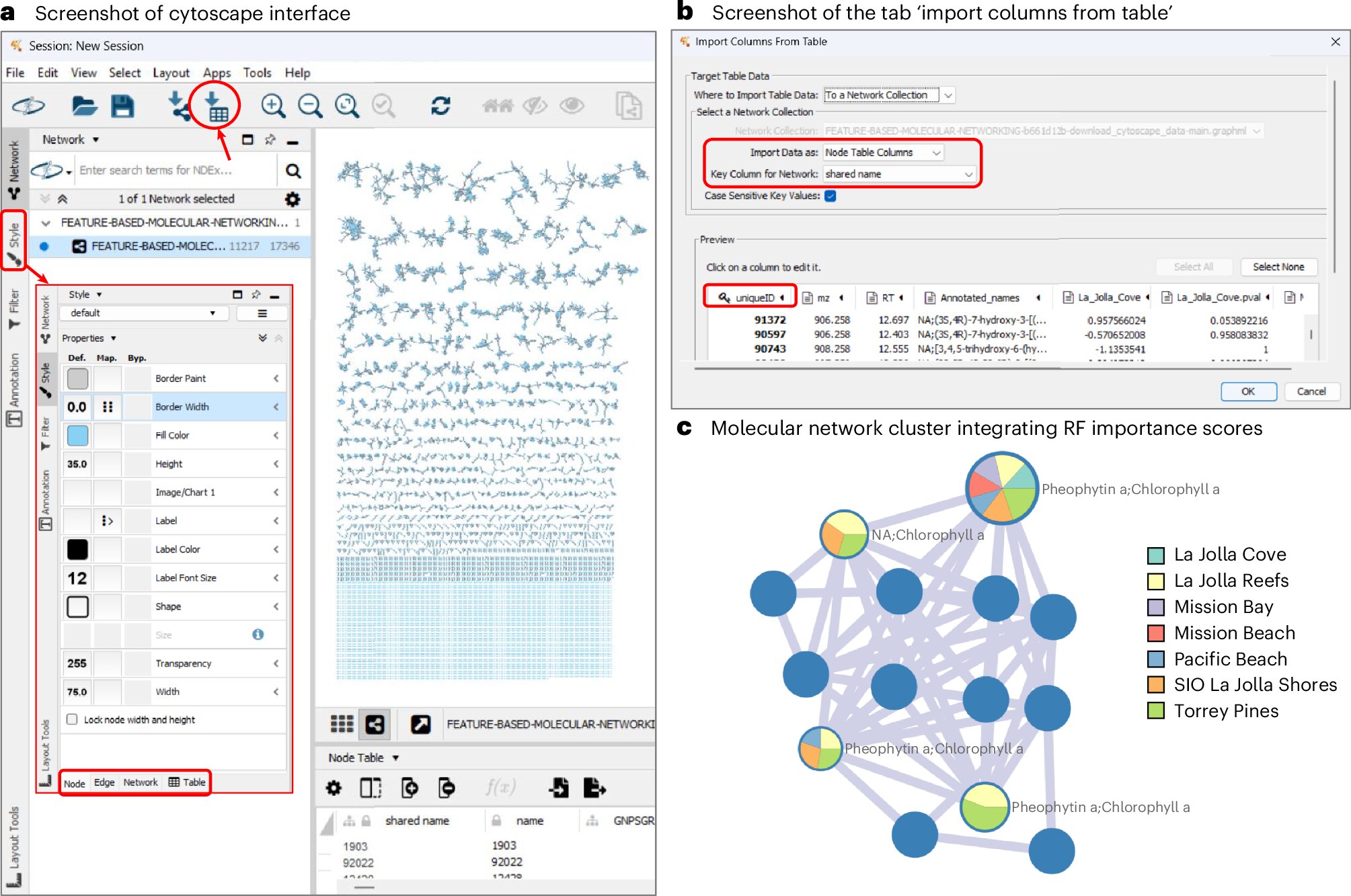
In jüngerer Zeit wandte das Team dieses neue Berechnungstool zur Analyse von Schadstoffen im Meerwasser Südkaliforniens an. Das Team erfasste schnell die chemischen Profile der Küstenumgebungen und zeigte mögliche Verschmutzungsquellen auf.
„Wir wollen verstehen, wie diese Schadstoffe in das Ökosystem gelangen“, sagt Daniel Petras, Assistenzprofessor für Biochemie an der University of California, Riverside, der das Forschungsteam leitete. „Aufgrund der großen chemischen Vielfalt des Ozeans ist es nicht einfach zu bestimmen, welche Moleküle im Ozean für die Umweltgesundheit wichtig sind. Das von uns entwickelte Protokoll beschleunigt diesen Prozess erheblich. Durch eine effizientere Datensortierung können wir Probleme im Zusammenhang mit der Meeresverschmutzung schneller verstehen. »
Petras und seine Kollegen berichten im Journal Naturprotokolle dass ihr Protokoll nicht nur für erfahrene Forscher, sondern auch für Bildungszwecke konzipiert ist, was es zu einer idealen Ressource für Studenten und Nachwuchswissenschaftler macht. Dieser rechnerische Arbeitsablauf wird von einer zugänglichen Webanwendung mit einer grafischen Benutzeroberfläche begleitet, die die Metabolomik-Datenanalyse auch für Laien zugänglich macht und es ihnen ermöglicht, innerhalb von Minuten statistische Erkenntnisse über ihre Daten zu gewinnen.
„Dieses Tool ist für ein breites Spektrum von Forschern zugänglich, vom Anfänger bis zum Experten, und ist für die Verwendung in Verbindung mit der molekularen Netzwerksoftware konzipiert, die meine Gruppe entwickelt“, sagte Mingxun Wang, Co-Autor und Assistenzprofessor für Informatik und Ingenieurwesen an der UCR. „Für Anfänger erleichtern die von uns bereitgestellten Richtlinien und Codes das Verständnis gängiger Datenverarbeitungs- und Analyseschritte. Für Experten beschleunigt es die reproduzierbare Datenanalyse und ermöglicht ihnen die gemeinsame Nutzung ihrer Arbeitsabläufe und Ergebnisse der statistischen Datenanalyse. »
Petras erklärte, dass die Forschungsarbeit einzigartig sei und als umfassende Bildungsressource fungiere, die von einer virtuellen Forschungsgruppe namens Virtual Multiomics Lab (VMOL) organisiert werde. Mit über 50 teilnehmenden Wissenschaftlern aus der ganzen Welt ist VMOL eine Open-Access-Community. Ziel ist es, den Prozess der chemischen Analyse zu vereinfachen und zu demokratisieren und ihn Forschern auf der ganzen Welt zugänglich zu machen, unabhängig von ihrem Hintergrund oder ihren Ressourcen.
„Ich bin äußerst stolz zu sehen, wie sich dieses Projekt zu etwas Wichtigem entwickelt hat, an dem Experten und Studenten aus der ganzen Welt beteiligt sind“, sagte Abzer Pakkir Shah, Doktorand in Petras‘ Gruppe und Erstautor des Artikels. „Durch die Beseitigung physischer und wirtschaftlicher Barrieren bietet VMOL Schulungen in computergestützter Massenspektrometrie und Datenwissenschaft an und zielt darauf ab, virtuelle Forschungsprojekte als eine neue Form der kollaborativen Wissenschaft zu starten. »
Die gesamte vom Team entwickelte Software ist kostenlos und öffentlich verfügbar. Die Entwicklung der Software wurde während einer Sommerschule zum Thema Untargeted Metabolomics im Jahr 2022 an der Universität Tübingen gestartet, wo das Team auch VMOL startete.
Petras hofft, dass das Protokoll besonders für Umweltforscher sowie für Wissenschaftler im biomedizinischen Bereich und für Forscher, die klinische Studien in der Mikrobiomwissenschaft durchführen, nützlich sein wird.
„Die Vielseitigkeit unseres Protokolls erstreckt sich auf ein breites Spektrum von Bereichen und Probentypen, einschließlich kombinatorischer Chemie, Dopinganalyse und Spurenkontamination von Lebensmitteln, Pharmazeutika und anderen Industrieprodukten“, sagte er.
Petras erlangte seinen Masterabschluss in Biotechnologie an der Hochschule Darmstadt und promovierte in Biochemie an der Technischen Universität Berlin. Er forschte als Postdoktorand an der UC San Diego, wo er sich auf die Entwicklung groß angelegter Methoden der Umweltmetabolomik konzentrierte. Im Jahr 2021 gründete er das Functional Metabolomics Laboratory an der Universität Tübingen. Im Januar 2024 wechselte er zur UCR, wo sich sein Labor auf die Entwicklung und Anwendung massenspektrometrischer Methoden zur Visualisierung und Bewertung des chemischen Austauschs innerhalb mikrobieller Gemeinschaften konzentriert.
Weitere Informationen:
Abzer K. Pakkir Shah et al., Statistische Analyse merkmalsbasierter molekularer Netzwerkergebnisse aus ungezielten Metabolomics-Daten, Naturprotokolle (2024). DOI: 10.1038/s41596-024-01046-3
Bereitgestellt von der University of California – Riverside
Zitat:Neues Data-Science-Tool beschleunigt die molekulare Analyse unserer Umwelt erheblich (2024, 20. September), abgerufen am 20. September 2024 von https://phys.org/news/2024-09-science-tool-greatly-molecular -analysis.html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient ausschließlich Informationszwecken.