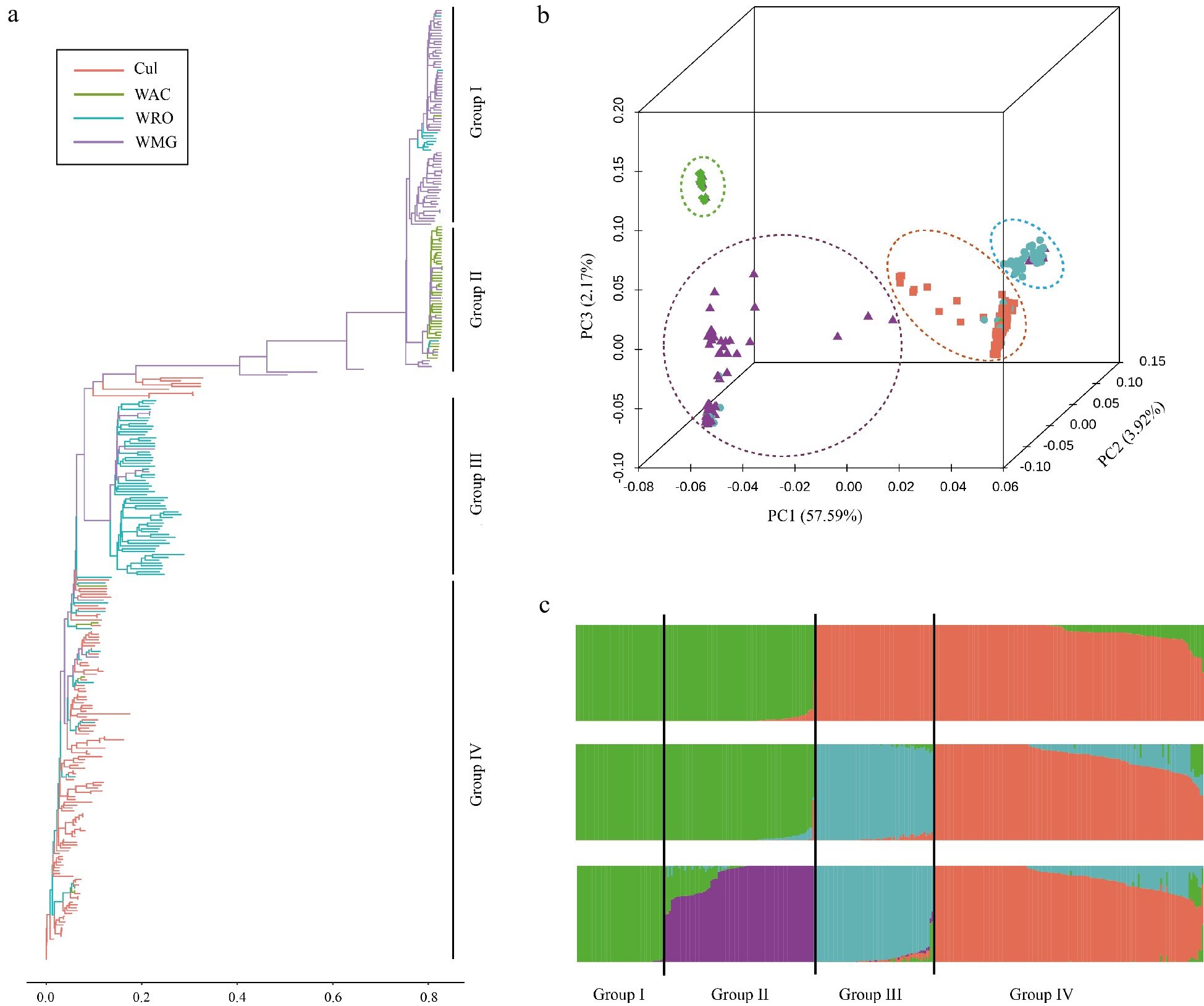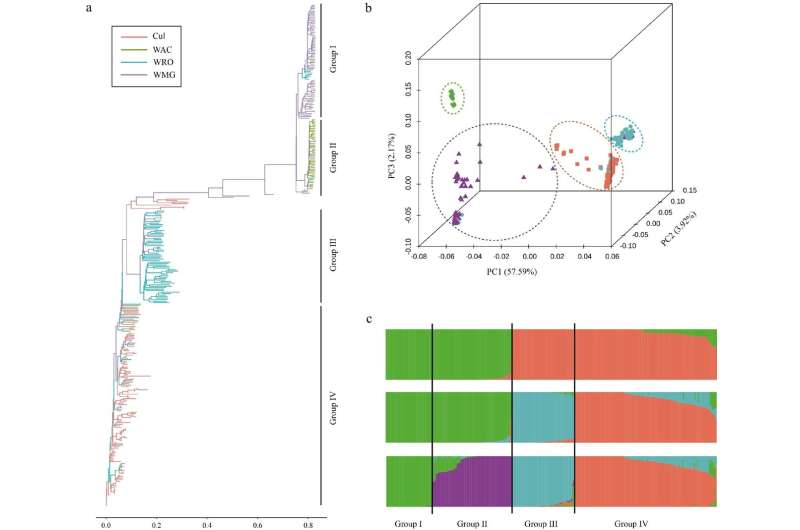
Anwendung auf die Analyse der genetischen Vielfalt von Populationen. (a) ADMIXTURE-Analyse. (b) Hauptkomponentenanalyse. (c) Strukturanalyse. Kredit : Tropische Pflanzen (2024). DOI: 10.48130/tp-0024-0020
Ein Forschungsteam entwickelte und validierte einen Single Nucleotide Polymorphism (SNP)-Flüssigkeitschip namens „HbGBTS80K“, der 80.080 SNPs enthält, die gleichmäßig auf 18 Chromosomen verteilt sind. Dieses SNP-Array unterschied effektiv 404 Kautschukakzessionen in vier Gruppen in der Analyse der genetischen Populationsdiversität und detektierte das Hauptgen HbPSK5 in GWAS für die Anzahl der Laticiferous-Ringe. Der HbGBTS80K-Chip ist ein wertvolles Werkzeug zur Beschleunigung funktioneller Studien und der molekularen Züchtung von Kautschukbäumen und überwindet die Ineffizienzen traditioneller Züchtungsmethoden.
Molekulare Marker sind spezifische DNA-Fragmente, die Unterschiede zwischen biologischen Individuen widerspiegeln und für die markergestützte Selektion unerlässlich sind. Während herkömmliche Marker wie RFLP, RAPD, AFLP und SSR eine begrenzte genomische Abdeckung aufweisen, sind SNPs in pflanzengenetischen Studien unverzichtbar geworden. Festphasen-SNP-Arrays verfügen über eine fortgeschrittene molekulare Züchtung, sind jedoch mit hohen Kosten und Einschränkungen bei der Fixierung von Zielorten konfrontiert. Flüssigphasenchips wie SNP-basierte GBTS bieten Flexibilität und Kosteneffizienz, werden jedoch in der Kautschukzüchtung nur unzureichend genutzt.
Eine Studie veröffentlicht in Tropische Pflanzen am 17. Mai 2024 zielt darauf ab, einen flüssigen SNP-Chip namens „HbGBTS80K“ zu entwickeln und zu validieren, um die Zuchteffizienz von Gummibäumen zu verbessern.
In dieser Studie wurde das HbGBTS80K-Array entworfen, indem zunächst das hochwertige Genom der Kautschuksorte „CATAS8-79“ zusammengesetzt und 335 Akzessionen in einer durchschnittlichen Tiefe von etwa 20x neu sequenziert wurden. Dieser Prozess erzeugte 5.323.701 SNPs, die auf der Grundlage der Häufigkeit kleiner Allele, der Deletionsrate und der Heterozygotie gefiltert wurden, um 96.044 SNPs für Einfangsonden auszuwählen.
Nach der Auswertung von 69 weiteren Akzessionen wurden 80.080 SNP-Standorte mit hoher Zuverlässigkeit beibehalten. Diese SNPs waren gleichmäßig über das Genom verteilt, mit der höchsten Dichte auf Chromosom 6 und der niedrigsten auf Chromosom 1. Die Genannotation ergab, dass 64,80 % der SNPs in der Körperregion des Gens lokalisiert waren, einschließlich exonischer, intronischer, stromaufwärts und stromabwärts liegender Region Bereiche.
Die ADMIXTURE-Analyse mit dem HbGBTS80K-Chip klassifizierte 404 Kautschukakzessionen in vier verschiedene Gruppen, was mit früheren Studien zur genetischen Vielfalt übereinstimmt. Der Chip zeigte auch eine hohe Genauigkeit in genomweiten Assoziationsstudien (GWAS), indem er das Hauptgen HbPSK5 für die Anzahl der Laticiferous Rings (NLR) identifizierte, das ein Schlüsselmerkmal für die Naturkautschukausbeute ist. Diese Validierung unterstreicht die Wirksamkeit des Chips sowohl bei der Analyse der genetischen Vielfalt als auch bei der Identifizierung funktioneller Gene und macht ihn zu einem wertvollen Werkzeug, um die molekulare Züchtung von Kautschukbäumen voranzutreiben.
Laut dem Hauptforscher der Studie, Weimin Tian, „ist der flüssige SNP-Chip HbGBTS80K ein wertvolles Werkzeug, das Funktionsstudien und die molekulare Züchtung von Gummibäumen erleichtern wird.“
Zusammenfassend umfasst das flüssige SNP-Array HbGBTS80K, das aus der Neusequenzierung des gesamten Genoms von 335 Gummiakzessionen entwickelt wurde, 80.080 SNPs, die auf 18 Chromosomen verteilt sind. Es unterscheidet effektiv Kautschukakzessionen in vier Gruppen und identifiziert genau das Hauptgen HbPSK5, das über GWAS mit Milchsäureringen assoziiert ist. Dieser Chip ist ein wertvolles Werkzeug zur Weiterentwicklung funktioneller Studien und der molekularen Selektion von Gummibäumen. In Zukunft wird der HbGBTS80K-Chip die Entwicklung hochwertiger Kautschuksorten erleichtern und als Modell für ähnliche Fortschritte in anderen Nutzpflanzen dienen.
Mehr Informationen:
Jinquan Chao et al., Design und Anwendung des HbGBTS80K-Flüssigkeitschips in Gummiholz, Tropische Pflanzen (2024). DOI: 10.48130/tp-0024-0020
Bereitgestellt von Maximum Academic Press
Zitat:Neuer flüssiger Einzelnukleotid-Polymorphismus-Chip kann die Zucht von Gummibäumen verbessern (2024, 22. Juli), abgerufen am 22. Juli 2024 von https://phys.org/news/2024-07-liquid-nucleotid-polymorphism-chip-rubber.html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient ausschließlich Informationszwecken.