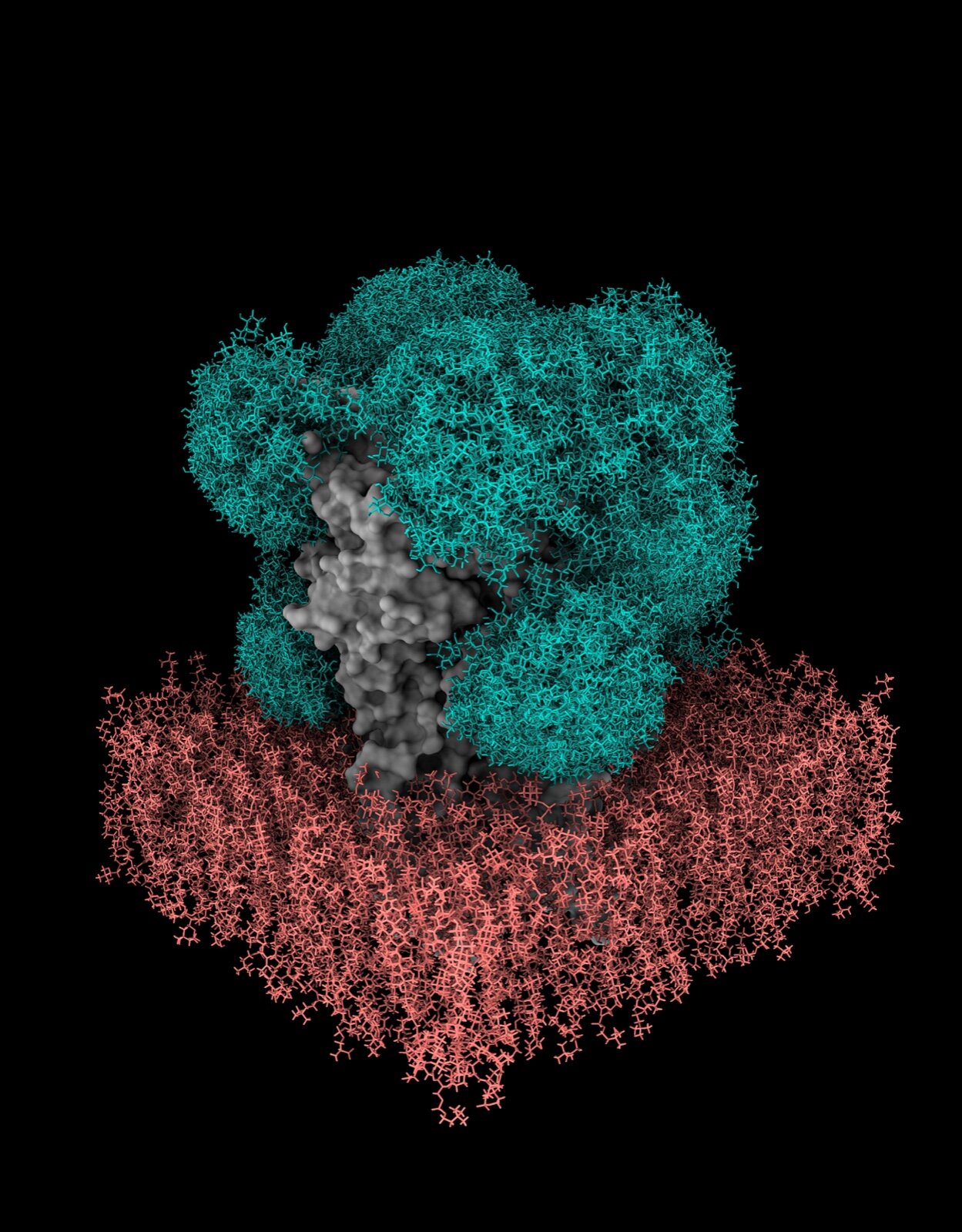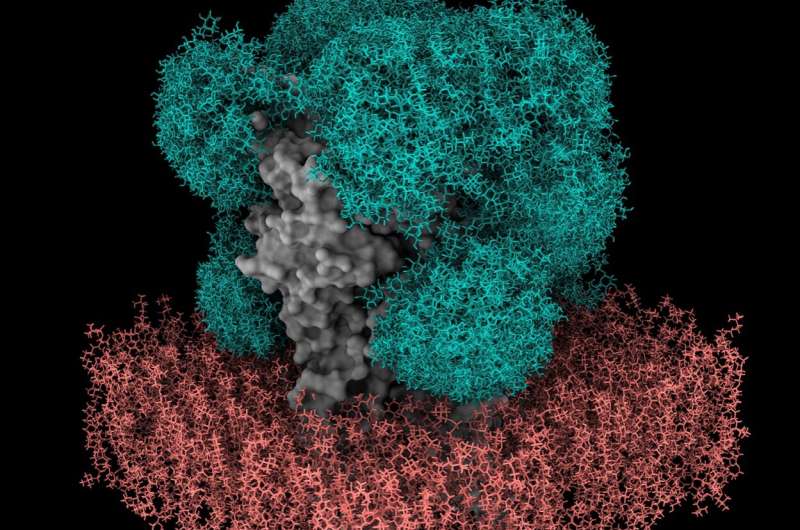
Modell des Zuckerschilds (grün) am GABAA-Rezeptor (grau) in einer von GlycoSHIELD erzeugten Membran (rot). Bildnachweis: Cyril Hanus, Inserm, Universität Paris-Cité
Proteine erfüllen nicht nur wesentliche Funktionen für das Überleben der Zellen, sondern beeinflussen auch die Entstehung und den Verlauf von Krankheiten. Um ihre Rolle bei Gesundheit und Krankheit zu verstehen, untersuchen Forscher die dreidimensionale Atomstruktur von Proteinen mithilfe experimenteller und rechnerischer Methoden.
Mehr als 75 Prozent der Proteine auf der Oberfläche unserer Zellen sind mit Glykanen bedeckt. Diese zuckerähnlichen Moleküle bilden sehr dynamische Schutzschilde um Proteine. Aufgrund der Mobilität und Variabilität von Zuckern ist es jedoch schwierig zu bestimmen, wie sich diese Schutzschilde verhalten oder wie sie die Bindung von Arzneimittelmolekülen beeinflussen.
Mateusz Sikora, Projektleiter und Direktor des Dioscuri Center for Modeling Post-Translational Modifications, und sein Team in Krakau sowie Partner am Max-Planck-Institut für Biophysik in Frankfurt am Main, Deutschland, haben diese Herausforderung mithilfe von Computern in Zusammenarbeit mit Wissenschaftlern festgestellt von Inserm in Paris, der Academia Sinica in Tapei und der Universität Bremen.
Ihr leistungsstarker neuer Algorithmus, GlycoSHIELD, ermöglicht eine schnelle, aber realistische Modellierung von Zuckerketten, die auf der Oberfläche von Proteinen vorhanden sind. Durch die Reduzierung der Rechenstunden und damit des Energieverbrauchs um Größenordnungen im Vergleich zu herkömmlichen Simulationstools ist GlycoSHIELD führend in Richtung Green Computing.
Die Arbeit wird in der Zeitschrift veröffentlicht Zelle.
Von Tausenden von Stunden bis zu wenigen Minuten
Schützende Glykanschilde beeinflussen stark die Art und Weise, wie Proteine mit anderen Molekülen, beispielsweise therapeutischen Medikamenten, interagieren. Beispielsweise verbirgt der Zuckerüberzug auf dem Spike-Protein des Coronavirus das Virus vor dem Immunsystem, indem es es natürlichen oder impfstoffinduzierten Antikörpern erschwert, das Virus zu erkennen.
Zuckerschilde spielen daher eine wichtige Rolle bei der Entwicklung von Medikamenten und Impfstoffen. Die pharmazeutische Forschung könnte von einer systematischen Vorhersage ihrer Morphologie und Dynamik profitieren. Doch die Vorhersage der Struktur von Zuckerschichten mithilfe von Computersimulationen war bisher nur mit fundierten Kenntnissen spezieller Supercomputer möglich. In vielen Fällen waren Tausende oder sogar Millionen Rechenstunden erforderlich.
Mit GlycoSHIELD bietet das Sikora-Team eine schnelle und umweltfreundliche Open-Source-Alternative. „Unser Ansatz reduziert die erforderlichen Ressourcen, Berechnungszeiten und technischen Fachkenntnisse“, erklärt Sikora.
„Jeder kann nun die Anordnung und Dynamik von Zuckermolekülen auf Proteinen in wenigen Minuten am Heimcomputer berechnen, ohne dass dafür spezielle Kenntnisse oder Hochleistungsrechner erforderlich sind. Darüber hinaus ist diese neue Art der Berechnung sehr energieeffizient.“ Die Software kann nicht nur in der Forschung eingesetzt werden, sondern könnte auch für die Entwicklung von Medikamenten oder Impfstoffen nützlich sein, beispielsweise in der Krebsimmuntherapie.
Ein Zuckerrätsel
Wie hat es das Team geschafft, eine solche Effizienzsteigerung zu erreichen? Die Autoren erstellten und analysierten eine Bibliothek mit Tausenden der wahrscheinlichsten 3D-Posen der häufigsten Formen von Zuckerketten auf Proteinen, die bei Menschen und Mikroorganismen vorkommen. Mithilfe langwieriger Simulationen und Experimente fanden sie heraus, dass es für eine zuverlässige Vorhersage der Glykan-Schutzschilde ausreicht, dass die angehängten Zucker nicht mit Membranen oder Teilen des Proteins kollidieren.
Der Algorithmus basiert auf diesen Ergebnissen. „GlyoSHIELD-Benutzer müssen lediglich das Protein und die Stellen angeben, an denen die Zucker gebunden sind. Unsere Software bringt sie dann in der wahrscheinlichsten Anordnung auf die Oberfläche des Proteins“, erklärt Sikora. „Wir konnten die Zuckerschilde des Spike-Proteins genau nachbilden: Sie sehen genauso aus wie das, was wir in Experimenten sehen.“
Mit GlycoSHIELD ist es nun möglich, neue und bestehende Proteinstrukturen mit Zuckerinformationen zu ergänzen. Die Wissenschaftler verwendeten GlycoSHIELD auch, um die Struktur von Zuckern am GABAA-Rezeptor aufzudecken, einem wichtigen Ziel für Beruhigungsmittel und Anästhetika.
Mehr Informationen:
Yu-Xi Tsai et al., Schnelle Simulation von Glykoproteinstrukturen durch Pfropfung und sterischen Ausschluss von Glykankonformerbibliotheken, Zelle (2024). DOI: 10.1016/j.cell.2024.01.034
Zur Verfügung gestellt von der Max-Planck-Gesellschaft
Zitat: Neue Software simuliert schnell Glykoproteinstrukturen, um die Arzneimittelentwicklung zu beschleunigen (1. März 2024), abgerufen am 1. März 2024 von https://phys.org/news/2024-03-software-rapidly-simulates-glycoprotein-drug.html
Dieses Dokument unterliegt dem Urheberrecht. Abgesehen von der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.