Der Nobelpreis für Chemie 2024 wurde an Demis Hassabis, John Jumper und David Baker für den Einsatz maschinellen Lernens zur Bewältigung einer der größten Herausforderungen der Biologie verliehen: die Vorhersage der 3D-Form von Proteinen und deren Entwurf von Grund auf.
Der diesjährige Preis zeichnet sich dadurch aus, dass er die Forschung eines Technologieunternehmens würdigt: DeepMind, ein KI-Forschungs-Startup, das 2014 von Google übernommen wurde. Die meisten bisherigen Chemie-Nobelpreise wurden an Universitätsforscher verliehen. Viele Gewinner haben daraufhin Start-ups gegründet, um ihre bahnbrechenden Arbeiten weiterzuentwickeln und zu kommerzialisieren (z. B. CRISPR-Genbearbeitungstechnologie und Quantenpunkte), aber die Forschung wurde von Anfang bis Ende nicht im kommerziellen Bereich durchgeführt.
Obwohl die Nobelpreise für Physik und Chemie getrennt vergeben werden, besteht im Jahr 2024 ein faszinierender Zusammenhang zwischen den Gewinnern der Forschung in diesen Bereichen. Der Physikpreis wurde an zwei Informatiker verliehen, die die Grundlagen des maschinellen Lernens legten, während die Gewinner der Chemie ausgezeichnet wurden Sie nutzen maschinelles Lernen, um eines der größten Rätsel der Biologie zu lösen: wie sich Proteine falten.
Die Nobelpreise 2024 unterstreichen sowohl die Bedeutung dieser Art künstlicher Intelligenz als auch die Tatsache, dass die heutige Wissenschaft häufig traditionelle Grenzen überschreitet und verschiedene Bereiche miteinander verbindet, um bahnbrechende Ergebnisse zu erzielen.
Die Herausforderung der Proteinfaltung
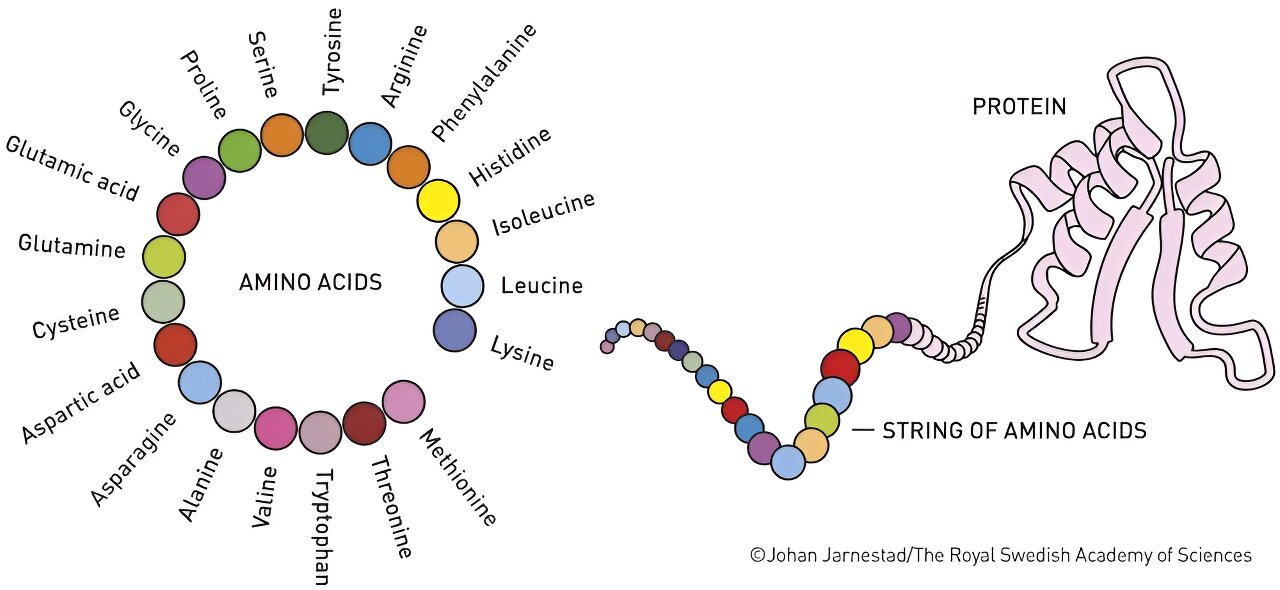
Proteine sind die molekularen Maschinen des Lebens. Sie bilden einen wichtigen Teil unseres Körpers, einschließlich Muskeln, Enzyme, Hormone, Blut, Haare und Knorpel.
Das Verständnis der Struktur von Proteinen ist wichtig, da ihre Form ihre Funktionen bestimmt. Im Jahr 1972 erhielt Christian Anfinsen den Nobelpreis für Chemie für den Nachweis, dass die Reihenfolge der Aminosäurebausteine eines Proteins die Form des Proteins bestimmt, was wiederum seine Funktion beeinflusst. Wenn sich ein Protein nicht richtig faltet, funktioniert es möglicherweise nicht richtig und kann zu Krankheiten wie Alzheimer, Mukoviszidose oder Diabetes führen.
Die Gesamtform eines Proteins hängt von den winzigen Wechselwirkungen, Anziehungskräften und Abstoßungen zwischen allen Atomen der Aminosäuren ab, aus denen es besteht. Manche wollen zusammen sein, andere nicht. Basierend auf mehreren Tausend dieser chemischen Wechselwirkungen verdreht und faltet sich das Protein in seine endgültige Form.
Jahrzehntelang bestand eine der größten Herausforderungen in der Biologie darin, die Form eines Proteins allein anhand seiner Aminosäuresequenz vorherzusagen. Obwohl Forscher jetzt ihre Form vorhersagen können, verstehen wir immer noch nicht, wie Proteine ihre spezifische Form annehmen und die Abstoßungen aller interatomaren Wechselwirkungen innerhalb von Mikrosekunden minimieren.
Um zu verstehen, wie Proteine funktionieren, und um Fehlfaltungen zu vermeiden, brauchten Wissenschaftler eine Möglichkeit, die Faltung von Proteinen vorherzusagen, aber die Lösung dieses Rätsels war keine leichte Aufgabe.
Im Jahr 2003 schrieb David Baker, ein Biochemiker an der University of Washington, Rosetta, ein Computerprogramm zum Design von Proteinen. Dadurch zeigte er, dass es möglich war, das Problem der Proteinfaltung umzukehren, indem man eine Proteinform entwarf und dann die zu ihrer Herstellung erforderliche Aminosäuresequenz vorhersagte.
Es war ein phänomenaler Fortschritt, aber die für die Berechnung gewählte Form war einfach und die Berechnungen komplex. Um konsequent neue Proteine mit den gewünschten Strukturen zu entwerfen, war ein großer Paradigmenwechsel erforderlich.
Eine neue Ära des maschinellen Lernens
Maschinelles Lernen ist eine Form der KI, bei der Computer lernen, Probleme durch die Analyse großer Datenmengen zu lösen. Es wird in verschiedenen Bereichen eingesetzt, von Spielen und Spracherkennung bis hin zu autonomen Fahrzeugen und wissenschaftlicher Forschung. Die Idee hinter maschinellem Lernen besteht darin, verborgene Muster in Daten zu nutzen, um komplexe Fragen zu beantworten.
Dieser Ansatz machte 2010 einen großen Schritt nach vorne, als Demis Hassabis DeepMind mitbegründete, ein Unternehmen, das sich zum Ziel gesetzt hat, Neurowissenschaften und KI zu kombinieren, um reale Probleme zu lösen.
Hassabis, ein Schachwunderkind im Alter von 4 Jahren, machte schnell Schlagzeilen mit AlphaZero, einer KI, die sich selbst beibrachte, Schach auf übermenschlichem Niveau zu spielen. Im Jahr 2017 schlug AlphaZero das weltbeste Computerschachprogramm Stockfish-8 deutlich. Die Fähigkeit der KI, aus ihrem eigenen Gameplay zu lernen, anstatt sich auf vorprogrammierte Strategien zu verlassen, markierte einen Wendepunkt in der Welt der KI.
Bald darauf wandte DeepMind ähnliche Techniken auf Go an, ein altes Brettspiel, das für seine immense Komplexität bekannt ist. Im Jahr 2016 besiegte sein KI-Programm AlphaGo einen der weltbesten Spieler, Lee Sedol, in einem weithin verfolgten Match, das Millionen von Zuschauern in Erstaunen versetzte.
Im Jahr 2016 verlagerte Hassabis den Fokus von DeepMind auf eine neue Herausforderung: das Problem der Proteinfaltung. Unter der Leitung von John Jumper, einem auf Proteinwissenschaften spezialisierten Chemiker, begann das AlphaFold-Projekt. Das Team nutzte eine große Datenbank experimentell ermittelter Proteinstrukturen, um die KI zu trainieren und ihr so das Erlernen der Prinzipien der Proteinfaltung zu ermöglichen. Das Ergebnis war AlphaFold2, eine KI, die in der Lage ist, die 3D-Struktur von Proteinen anhand ihrer Aminosäuresequenzen mit bemerkenswerter Genauigkeit vorherzusagen.
Dies war ein wichtiger wissenschaftlicher Durchbruch. AlphaFold hat seitdem die Strukturen von mehr als 200 Millionen Proteinen vorhergesagt, im Wesentlichen alle Proteine, die Wissenschaftler bisher sequenziert haben. Diese umfangreiche Datenbank mit Proteinstrukturen ist jetzt frei verfügbar und beschleunigt die Forschung in Biologie, Medizin und Arzneimittelentwicklung.
Synthetische Proteine zur Bekämpfung von Krankheiten
Das Verständnis der Faltung und Funktion von Proteinen ist für die Entwicklung neuer Medikamente von entscheidender Bedeutung. Enzyme, eine Proteinart, wirken als Katalysatoren bei biochemischen Reaktionen und können diese Prozesse beschleunigen oder regulieren. Um Krankheiten wie Krebs oder Diabetes zu behandeln, zielen Forscher häufig auf bestimmte Enzyme ab, die am Krankheitsverlauf beteiligt sind. Durch die Vorhersage der Form eines Proteins können Wissenschaftler bestimmen, wo kleine Moleküle – potenzielle Arzneimittelkandidaten – daran binden könnten. Dies ist der erste Schritt bei der Entwicklung neuer Arzneimittel.
Im Jahr 2024 brachte DeepMind AlphaFold3 auf den Markt, eine verbesserte Version des AlphaFold-Programms, das nicht nur die Form von Proteinen vorhersagt, sondern auch potenzielle Bindungsstellen für kleine Moleküle identifiziert. Dieser Fortschritt erleichtert es Forschern, Medikamente zu entwickeln, die genau auf die richtigen Proteine abzielen.
Google kaufte Deepmind im Jahr 2014 für rund eine halbe Milliarde Dollar. Google DeepMind hat nun ein neues Unternehmen, Isomorphic Labs, gegründet, um mit Pharmaunternehmen zusammenzuarbeiten, um mithilfe dieser AlphaFold3-Vorhersagen echte Medikamente zu entwickeln.
David Baker seinerseits leistete weiterhin wichtige Beiträge zur Proteinwissenschaft. Sein Team an der University of Washington entwickelte eine KI-basierte Methode namens „Familienhalluzination“, mit der sie völlig neue Proteine von Grund auf entwerfen konnten. Halluzinationen sind neue Muster – in diesem Fall Proteine –, die plausibel sind, also gut mit den Mustern in den KI-Trainingsdaten übereinstimmen. Zu diesen neuen Proteinen gehörte ein lichtemittierendes Enzym, was zeigt, dass maschinelles Lernen bei der Schaffung neuer synthetischer Proteine helfen kann. Diese KI-Tools bieten neue Möglichkeiten zur Entwicklung funktioneller Enzyme und anderer Proteine, die sich auf natürliche Weise nie hätten entwickeln können.
KI wird das nächste Kapitel der Forschung aufschlagen
Die nobelpreiswürdigen Leistungen von Hassabis, Jumper und Baker zeigen, dass maschinelles Lernen nicht nur ein Werkzeug für Informatiker ist: Es ist heute ein wesentlicher Bestandteil der Zukunft von Biologie und Medizin.
Durch die Bewältigung eines der anspruchsvollsten Probleme der Biologie haben die Preisträger des Jahres 2024 neue Möglichkeiten in der Arzneimittelforschung, der personalisierten Medizin und sogar unserem Verständnis der Chemie des Lebens selbst eröffnet.
Bereitgestellt von The Conversation
Dieser Artikel wurde von The Conversation unter einer Creative Commons-Lizenz erneut veröffentlicht. Lesen Sie den Originalartikel.![]()
Zitat: Maschinelles Lernen löste das Proteinfaltungsproblem und gewann den Nobelpreis für Chemie 2024 (12. Oktober 2024), abgerufen am 12. Oktober 2024 von https://phys.org/news/2024-10-machine-protein -problem-won- nobel.html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient ausschließlich Informationszwecken.