Kredit: Nukleinsäureforschung (2024). DOI: 10.1093/nar/gkae689
DNA, das Molekül, das die genetische Information aller lebenden Organismen trägt, ist auf komplexe Weise in Zellen verpackt, die eine effiziente Funktion ermöglichen. Nukleosomen erleichtern die DNA-Verdichtung und spielen auch eine entscheidende Rolle bei der Regulierung der Genexpression und anderer biologischer Prozesse.
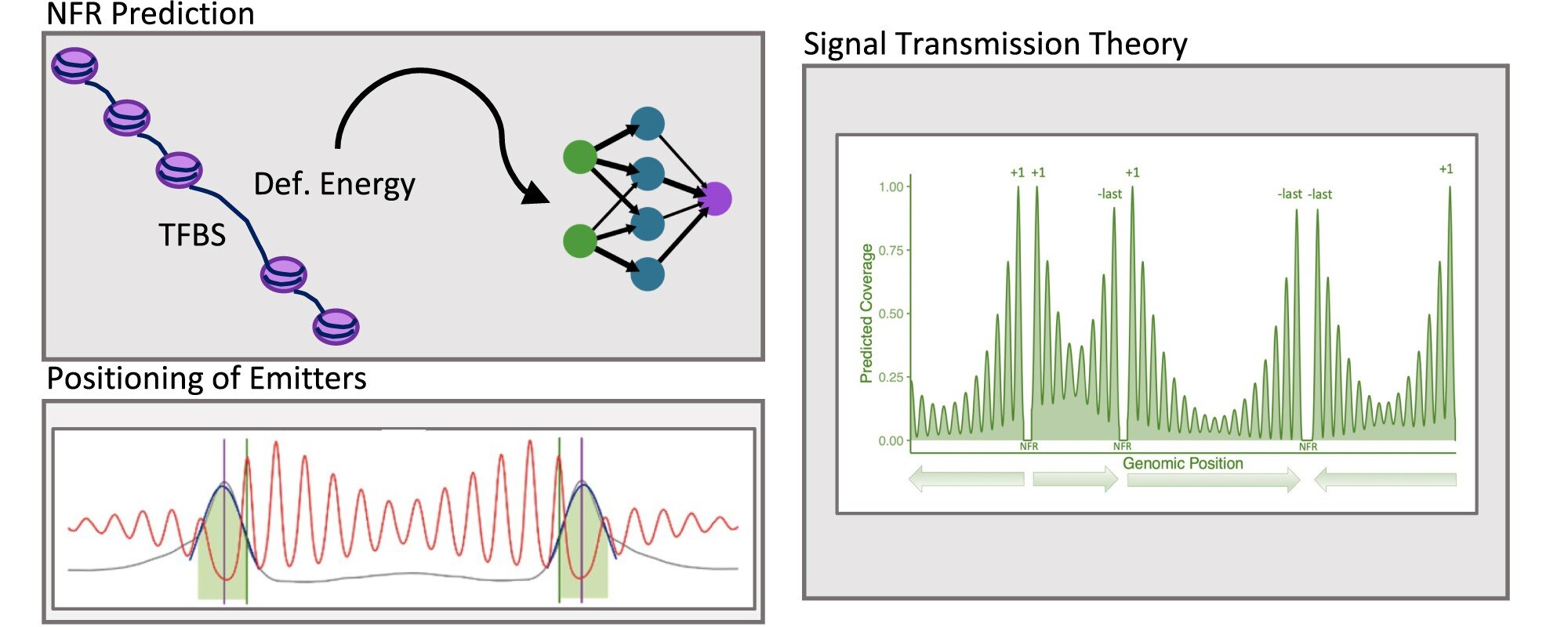
Ein Team von Wissenschaftlern unter der Leitung von Dr. Modesto Orozco vom IRB Barcelona hat eine fortschrittliche Rechentechnik entwickelt, um die Genarchitektur anhand der Position von Nukleosomen vorherzusagen. Die Methode kombiniert experimentelle Ansätze mit Techniken des maschinellen Lernens und der Signalübertragungstheorie. Die Studie wurde in der Fachzeitschrift veröffentlicht Nukleinsäureforschung.
Ein Vorhersagemodell, das experimentellen Methoden Konkurrenz macht
In den letzten Jahren haben Wissenschaftler experimentelle Techniken wie MNase-seq verwendet, um Nukleosomen zu kartieren. Das von Dr. Orozcos Team entwickelte Modell nutzt Informationen über DNA-Sequenzen und physikalische Eigenschaften nicht nur, um experimentelle Daten zu reproduzieren, sondern auch, um die Position von Nukleosomen schneller und genauer vorherzusagen.
„Die Genauigkeit unseres Modells ist mit der der fortschrittlichsten experimentellen Methoden vergleichbar“, sagt Dr. Orozco, Leiter des Labors für molekulare Modellierung und Bioinformatik am IRB Barcelona und ordentlicher Professor an der Universität Barcelona.
Implikationen für Genregulation und Biomedizin
Die Studie zeigt, dass die nukleosomale Architektur stark von der DNA-Sequenz und den von den Genenden ausgesendeten physikalischen Signalen beeinflusst wird. Diese Signale bestimmen die Position des ersten und letzten Nukleosoms (+1 und -letztes) und beeinflussen auch die Position der Nukleosomen entlang des Gens.
„Unsere Arbeit legt nahe, dass die Struktur von Nukleosomen die Genexpression auf komplexere Weise beeinflussen könnte, als wir dachten“, fügt Alba Sala, Doktorandin am IRB Barcelona und Erstautorin der Studie, hinzu.
Dieser Ansatz ist für zukünftige Forschungen darüber, wie Veränderungen in der Chromatinstruktur den Krankheitsausbruch beeinflussen können, von entscheidender Bedeutung. Durch ein besseres Verständnis der Organisation von DNA und Nukleosomen können Wissenschaftler neue therapeutische Ziele identifizieren und wirksamere Behandlungen entwickeln.
Weitere Informationen:
Alba Sala et al., Ein integriertes maschinelles Lernmodell zur Vorhersage der Nukleosomenarchitektur, Nukleinsäureforschung (2024). DOI: 10.1093/nar/gkae689
Bereitgestellt vom Biomedicine Research Institute (IRB Barcelona)
Zitat: Erweitertes Modell sagt Genarchitektur über Nukleosomenposition voraus (10. Oktober 2024), abgerufen am 10. Oktober 2024 von https://phys.org/news/2024-10-advanced-gene-architecture-nucleosome-position html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient ausschließlich Informationszwecken.