Kredit: Nukleinsäureforschung (2024). DOI: 10.1093/nar/gkae780
Die meiotische Rekombination erzeugt genetische Vielfalt und fördert die ordnungsgemäße Chromosomentrennung der Elternchromosomen. Dieser Prozess erfordert, dass eine Reihe von Rekombinasen, die auf einzelsträngigen (ss) DNAs, sogenannten Nukleoproteinfilamenten, polymerisiert sind, einer Homologiesuche und einem Strangaustausch zwischen homologen DNAs unterzogen werden.
Bei der Meiose von Saccharomyces cerevisiae werden von Spo11 programmierte DNA-Doppelstrangbrüche (DSBs) gebildet, um 3′-ssDNA-Schwänze zu erzeugen. Einmal gebildet, werden ssDNA-Überhänge schnell durch das reichlich vorhandene hochaffine ssDNA-bindende Protein, das Replikationsprotein A (RPA), gebunden, um diese ssDNAs vor nukleolytischem Abbau oder der Bildung von DNA höherer Ordnung zu schützen.
RPA-beschichtete ssDNA-Substrate unterscheiden sich von reinen ssDNA-Substraten aufgrund der hohen Affinität von RPA für ssDNA; Daher ist der Rekombinationsmediator-Proteinkomplex Mei5-Sae3 für die Bindung von Rekombinasen an RPA-beschichteter ssDNA erforderlich. Die mechanistische Rolle von Mei5-Sae3 bei der Vermittlung der Dmc1-Aktivität bleibt jedoch unklar.
Um zu untersuchen, wie Mei5-Sae3 Dmc1 dazu anregt, RPA zu bewegen und Nukleoproteinfilamente zu bilden, verwendete das Forschungsteam von NTU Chemical, NTU IBS und der Universität Osaka biochemische Proteinreinigungstechniken und FRET-Einzelmolekül- und Kolokalisations-Einzelmolekülspektroskopie (CoSMoS)-Techniken zur Erfassung Echtzeitbindung von Dmc1 und Dissoziation von RPA auf individueller DNA mit außergewöhnlicher zeitlicher Auflösung.
Ihre Ergebnisse werden in der Zeitschrift veröffentlicht Nukleinsäureforschung.
Im Gegensatz zu herkömmlichen biologischen Ansätzen, die in erster Linie die endgültigen Gleichgewichtsprodukte von Ensemblereaktionen untersuchen, könnten Einzelmolekülmethoden die Beiträge einzelner biochemischer Schritte einzelner Moleküle aufklären und so vorübergehende Zwischenzustände aufdecken, die Einblick in den Reaktionsverlauf geben könnten.
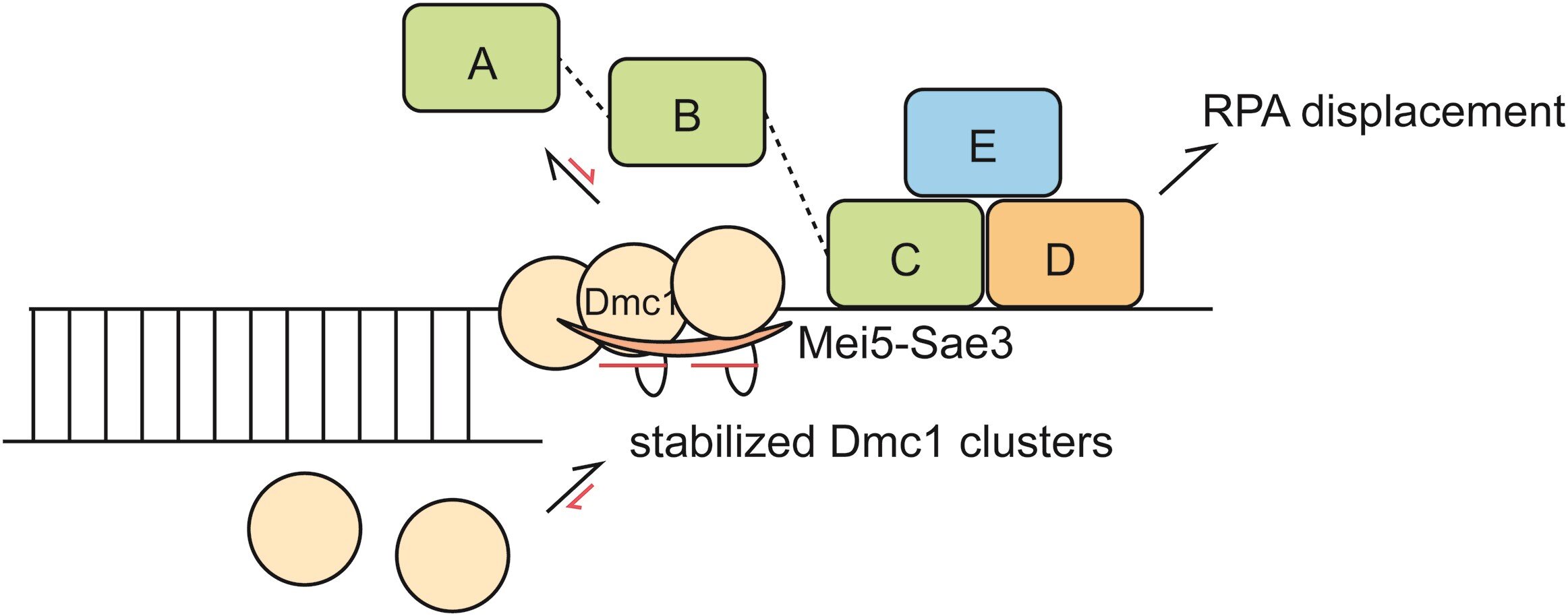
Das Ergebnis zeigte, dass Mei5-Sae3 Dmc1-Keimbildungscluster mit 2–3 Molekülen auf nackter DNA stabilisierte, indem es die Dmc1-Dissoziationsraten bevorzugt reduzierte. Mei5-Sae3 stimulierte auch die Dmc1-Assemblierung auf RPA-beschichteter DNA.
Unter Verwendung von GFP-markiertem RPA wurde die Koexistenz eines Zwischenprodukts mit Dmc1 und RPA auf ssDNA vor der RPA-Dissoziation beobachtet. Darüber hinaus hing die Verdrängungseffizienz von RPA von der Dmc1-Konzentration ab und ihre Abhängigkeit korrelierte positiv mit der Stabilität von Dmc1-Clustern auf kurzer ssDNA.
Diese Ergebnisse legen ein molekulares Modell nahe, dass Mei5-Sae3 die Bindung von Dmc1 an RPA-beschichteter ssDNA vermittelt, indem es Dmc1-Keimbildungscluster stabilisiert und dadurch die Dynamik von RPA auf DNA beeinflusst, um die RPA-Dissoziation zu fördern.
Die Forschung trägt zum ersten detaillierten molekularen Modell bei, das jemals für dieses einzigartige Mediatorprotein Mei5-Sae3 veröffentlicht wurde, und erklärt, wie eine Mediator-Rekombinase-Wechselwirkung die Rekombinase-Assemblierung auf RPA-beschichteter ssDNA stimulieren kann.
Weitere Informationen:
Chin-Dian Wei et al., Mei5–Sae3 stabilisiert Dmc1-Keimbildungscluster für den effizienten Aufbau von Dmc1 auf RPA-beschichteter einzelsträngiger DNA, Nukleinsäureforschung (2024). DOI: 10.1093/nar/gkae780
Zur Verfügung gestellt von der National Taiwan University
Zitat: Forscher enthüllen Schlüsselmechanismus bei der Regulierung der DNA-Rekombination (4. Oktober 2024), abgerufen am 5. Oktober 2024 von https://phys.org/news/2024-10-reveal-key-mechanism-dna-recombination.html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient ausschließlich Informationszwecken.