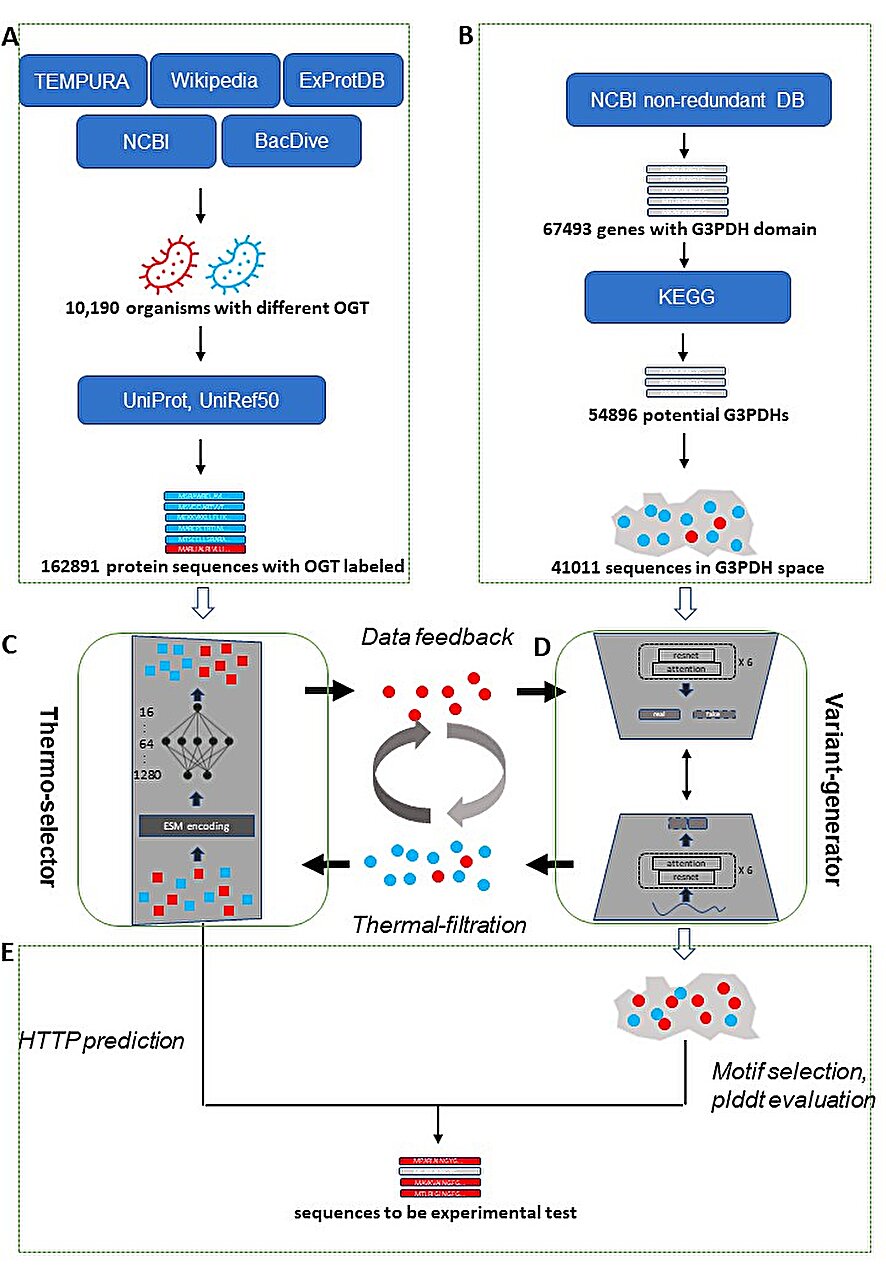
Schematisches Diagramm, das das Framework und den Datenfluss von DeepEvo darstellt. Bildnachweis: Huanyu Chu et al.
Die Schaffung von Proteinen zur Erzielung wünschenswerter Eigenschaften ist der heilige Gral der modernen Biotechnologie. Beispielsweise kann die Lebensmittelindustrie von künstlichen Enzymen profitieren, die im Vergleich zu natürlichen Enzymen biochemische Reaktionen bei höheren Temperaturen verstärken können. Dadurch können wertvolle Ressourcen wie Arbeit, Geld und Zeit eingespart werden. Der Prozess, zu einem funktionellen Protein von Interesse mit der gewünschten Eigenschaft zu gelangen, stellt jedoch erhebliche Herausforderungen dar.
Aktuelle Ansätze des Protein-Engineerings, wie beispielsweise die gerichtete Evolution, basieren weitgehend auf dem Zufall, um ideale Varianten des interessierenden Proteins einzugrenzen. Die gerichtete Evolution nutzt die wiederholte Einführung von Proteinsequenzänderungen, die als Mutationen bezeichnet werden (iterative Mutagenese), gefolgt von einem schnellen Screening einer großen Anzahl unterschiedlicher Proteine (Hochdurchsatz-Screening). Es ist nicht verwunderlich, dass diese Methode mühsam und ineffektiv ist.
Um diese Einschränkungen zu überwinden, entwickelte eine Gruppe chinesischer Forscher unter der Leitung von Dr. Huifeng Jiang vom Tianjin Institute of Industrial Biotechnology der Chinesischen Akademie der Wissenschaften und dem National Technological Innovation Center for Synthetic Biology eine Strategie für KI-basiertes Protein-Engineering namens „ DeepEvo“.
Weiter erklärt Dr. Jiang: „DeepEvo verwendet eine Deep-Evolution-Strategie, die die Prinzipien des Deep Learning (ein Prozess, der die Funktionsweise des lebenden Gehirns nachahmt) und der Evolutionsbiologie kombiniert. » Die Studie wurde online veröffentlicht in BioDesign-Forschung am 20. März 2024.
Forscher nutzten DeepEvo, um die Hochtemperaturtoleranz eines Enzyms namens Glycerinaldehyd-3-phosphat-Dehydrogenase (G3PDH) zu verbessern. G3PDH spaltet Glukose auf, um während der Glykolyse in lebenden Zellen Energie zu erzeugen. Als das Team die Ergebnisse von DeepEvo experimentell validierte, erreichte es eine vielversprechende Erfolgsquote von mehr als 26 %.
In der Studie umfassten die für DeepEvo verwendeten Daten Sequenzen von Organismen mit unterschiedlichen optimalen Wachstumstemperaturen (OGT) und natürliche Sequenzen mit den gewünschten Funktionen. Die entwickelte DeepEvo-Strategie umfasste einen Selektor (Thermo-Selektor) und einen Variantengenerator (Variant-Generator), um funktionelle Proteinsequenzen zu erzeugen, die das gewünschte Merkmal enthalten.
Während der Selektor als selektiver Druck zur Anreicherung der gewünschten Proteinsequenzen fungierte, produzierte der Variantengenerator diese Sequenzen, in diesem Fall G3PDH-Varianten mit hoher Temperaturtoleranz. Mit OGT markierte Sequenzen trainierten den Thermoselektor, während Sequenzen mit der gewünschten Funktion den Variantengenerator trainierten. Der Thermoselektor filterte die Sequenzen und steuerte den Variantengenerator.
Bemerkenswerterweise bildete ein Protein-Sprachmodell – eine Art Deep-Learning-Modell – die Grundlage des in dieser Studie verwendeten Thermoselektors. Diese Modelle werden anhand großer Mengen realer Proteinsequenzdaten trainiert, um die diesen Sequenzen innewohnenden Muster und Merkmale zu lernen. Dieser entwickelte Selektor verwendet eine erlernte Darstellung von Proteinsequenzen, um die Generierung und Auswahl von Sequenzen zu steuern, die das gewünschte Merkmal aufweisen.
Darüber hinaus sammelten die Forscher durch einen iterativen Prozess, an dem der Generator und der Selektor beteiligt waren, Merkmale einer hohen Temperaturtoleranz in den Proteinsequenzen. Die iterative Verfeinerung von Sequenzen, von denen vorhergesagt wurde, dass sie hohen Temperaturen standhalten, bildete einen Sequenzgenerierungszyklus.
Darüber hinaus fügt Dr. Jiang hinzu: „Der iterative Prozess in DeepEvo ahmt den Prozess der natürlichen Selektion nach, bei dem funktionelle Sequenzen über aufeinanderfolgende Generationen bevorzugt und akkumuliert werden, was letztendlich zur Entwicklung von Proteinvarianten mit gewünschten Eigenschaften führt.“ »
Daher verifizierten die Forscher die vorhergesagten hochtemperaturtoleranten Proteinsequenzen und behielten die funktionellen Motive durch Nasslaborexperimente bei. Aus den 30 generierten Sequenzen erhielten sie acht Varianten, was die hohe Effizienz von DeepEvo bei der Entwicklung hochtemperaturtoleranter Proteine unterstreicht.
In Zukunft könnte DeepEvo die Auswahl verschiedener interessanter Merkmale erleichtern, nicht nur der hohen Temperaturtoleranz. In diesem Zusammenhang bemerkt Dr. Jiang: „Wir könnten den DeepEvo-Ansatz anwenden, um andere Proteineigenschaften wie Säure-Base-Toleranz, katalytische Aktivität und Antigenaffinität zu manipulieren und so die Erzeugung neuer Proteine mit mehreren gewünschten Eigenschaften zu erleichtern.“
Dank der Bemühungen von Dr. Jiang und seiner Forschungsgruppe hat DeepEvo somit den Weg für effizientes Protein-Engineering geebnet. Eine einfache und effiziente Produktion von Proteinen, die auf die gewünschten Eigenschaften zugeschnitten sind, könnte bald Realität werden.
Mehr Informationen:
Huanyu Chu et al., Entwicklung hochtemperaturtoleranter Proteine durch tiefe Evolution, Forschung im BioDesign (2024). DOI: 10.34133/bdr.0031
Bereitgestellt von der NanJing Agricultural University
Zitat: DeepEvo: Eine „intelligente“ Strategie für personalisiertes Protein-Engineering (27. Juni 2024), abgerufen am 27. Juni 2024 von https://phys.org/news/2024-06-deepevo-intelligent-strategy-customized-proteins html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.