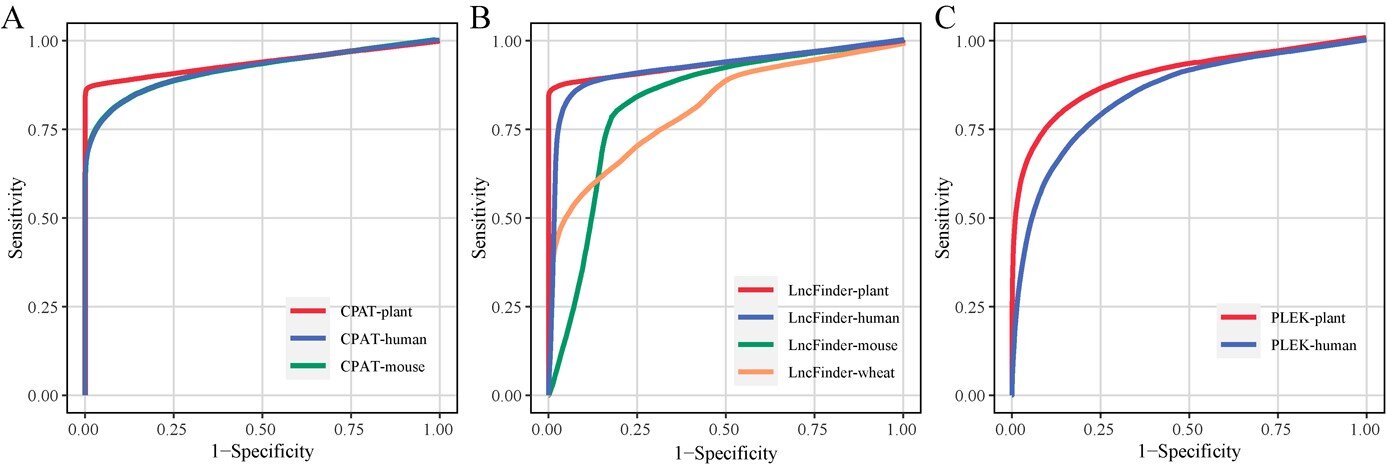
ROC-Kurven von neu trainierten und ursprünglichen Modellen anhand von Datensätzen von 20 Pflanzenarten. Eine ROC-Kurve des neu trainierten CPAT-Pflanzenmodells und ihr Vergleich mit den ursprünglichen Menschen- und Mausmodellen. B ROC-Kurven des recycelten LncFinder-Pflanzenmodells und Vergleich mit den ursprünglichen Menschen-, Maus- und Weizenmodellen. C ROC-Kurven des recycelten PLEK-Pflanzenmodells und sein Vergleich mit dem ursprünglichen menschlichen Modell. Kredit: Gartenbauforschung (2024). DOI: 10.1093/hr/uhae041
Lange nichtkodierende RNAs (lncRNAs) sind allgegenwärtige Transkripte, die eine entscheidende regulatorische Rolle in verschiedenen biologischen Prozessen spielen, darunter Chromatin-Remodellierung, posttranskriptionelle Regulation und epigenetische Modifikationen. Obwohl immer mehr Beweise die Mechanismen aufklären, durch die pflanzliche ncRNAs Wachstum, Wurzelentwicklung und Samenruhe modulieren, bleibt ihre genaue Identifizierung aufgrund des Mangels an pflanzenspezifischen Methoden schwierig.
Derzeit werden traditionelle Methoden zur Identifizierung pflanzlicher lncRNAs größtenteils auf der Grundlage menschlicher oder tierischer Datensätze entwickelt. Daher wurden die Genauigkeit und Effizienz dieser Methoden zur Vorhersage pflanzlicher ncRNAs noch nicht vollständig bewertet.
Kürzlich wurde in veröffentlicht Gartenbauforschung.
Diese Studie sammelte umfassend hochwertige RNA-Sequenzierungsdaten aus verschiedenen Pflanzen und nutzte diese pflanzenspezifischen Daten, um Modelle von drei traditionellen lncRNA-Vorhersagetools, nämlich CPAT, LncFinder und PLEK, neu zu trainieren. Die Leistung der neu trainierten Modelle wurde mit anderen gängigen lncRNA-Vorhersagetools wie CPC2, CNCI, RNAplonc und LncADeep verglichen und bewertet.
Die Ergebnisse zeigten, dass die neu trainierten Modelle die Vorhersageleistung pflanzlicher ncRNAs deutlich verbesserten. Unter ihnen übertrafen zwei neu trainierte Modelle, LncFinder-plant und CPAT-plant, andere bei mehreren Bewertungsmetriken, was sie zu den am besten geeigneten Werkzeugen für die Identifizierung pflanzlicher lncRNA macht.
Diese Forschung entwickelte eine Computerpipeline namens Plant-LncPipe zur Identifizierung und Analyse pflanzlicher lncRNAs.
Diese Pipeline integriert zwei leistungsstärkste Identifizierungsmodelle, CPAT-plant und LncFinder-plant, und ermöglicht einen vollständigen Berechnungsprozess, der die Vorverarbeitung von Rohdaten, die Assemblierung von Transkripten, die lncRNA-Identifizierung, die lncRNA-Klassifizierung und die Herkunft von lncRNAs umfasst. Diese rechnerische Pipeline kann in großem Umfang auf verschiedene Pflanzenarten angewendet werden. Plant-LncPipe ist öffentlich verfügbar.
Die Studie zeigt, dass die Neuschulung von lncRNA-Vorhersagemodellen anhand hochwertiger transkriptomischer Daten von Pflanzen eine genauere Erfassung pflanzlicher lncRNA-Merkmale ermöglichte, was die Genauigkeit und Zuverlässigkeit der Vorhersagen erheblich verbesserte. Die Studie betonte die Bedeutung einer artspezifischen Umschulung zur Verbesserung der Modellgenauigkeit. Durch die Wiederverwendung vorhandener ausgereifter Modelle wurden zuvor gesammelte Erfahrungen und Methoden beibehalten und gleichzeitig die Anwendbarkeit und Genauigkeit der Modelle weiter verbessert.
Mehr Informationen:
Xue-Chan Tian et al, Plant-LncPipe: eine rechnerische Pipeline, die eine erhebliche Verbesserung bei der Identifizierung pflanzlicher lncRNA bietet, Gartenbauforschung (2024). DOI: 10.1093/hr/uhae041
Zur Verfügung gestellt von der Chinesischen Akademie der Wissenschaften
Zitat: Ein neues Tool zur Identifizierung langer nicht-kodierender RNAs in Pflanzen (1. Mai 2024), abgerufen am 1. Mai 2024 von https://phys.org/news/2024-05-tool-coding-rna-identification.html
Dieses Dokument unterliegt dem Urheberrecht. Abgesehen von der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.