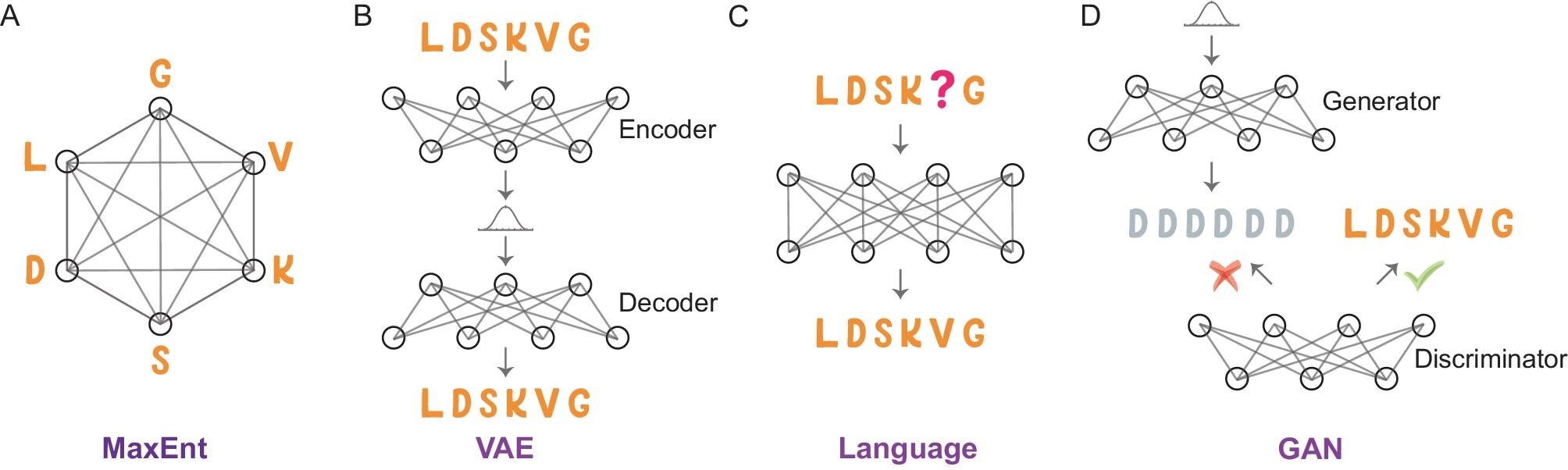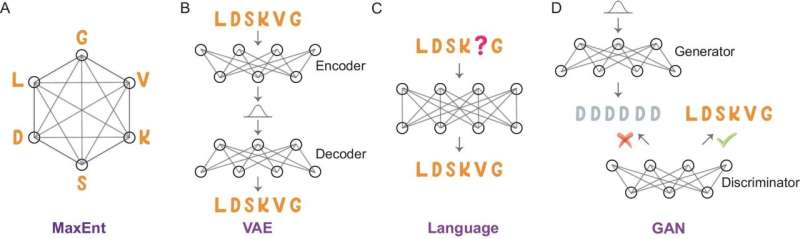
Vergleichende Darstellung generativer Modelle zur Modellierung von Proteinsequenzen. (A) MaxEnt-Modell: Dieses Modell zielt darauf ab, sowohl die Erhaltung einzelner Aminosäuren als auch ihre paarweisen Wechselwirkungen abzugrenzen und gleichzeitig minimale Annahmen zu treffen, indem die Entropie der Sequenzinformationen maximiert wird. (B) VAE: ein neuronales Netzwerk, das lernt, Daten in einen niedrigerdimensionalen latenten Raum zu kodieren und sie dann zu dekodieren; Nach dem Training können effizient neue Daten generiert werden, die dem Trainingssatz ähneln. (C) Sprachmodell: Ein maskiertes Sprachmodell verwendet einen vorhersagebasierten Mechanismus, der darauf abzielt, die maskierte Aminosäure genau vorherzusagen und so die Verteilung eines Korpus von Proteinsequenzen zu lernen. (D) GAN: Ein Framework verwendet zwei zusammenarbeitende neuronale Netze: einen Generator, der neue Proteinsequenzen erstellt, und einen Diskriminator, der ihre Authentizität bewertet. Kredit: Nationale wissenschaftliche Zeitschrift (2023). DOI: 10.1093/nsr/nwad331
Enzyme, die bemerkenswerten Biokatalysatoren der Natur, spielen in verschiedenen Aspekten des täglichen Lebens eine wesentliche Rolle. Betrachten Sie das beeindruckende Schauspiel der Glühwürmchen, die eine Sommernacht erhellen. Ihr bezaubernder Glanz verzaubert nicht nur den Betrachter, sondern spielt auch eine Rolle bei der Kommunikation und Paarung. Dieses Naturschauspiel wird durch ein Enzym namens Luciferase angetrieben.
Auf molekularer Ebene sind Enzyme komplexe Netzwerke aus Aminosäuren. Luciferase, ein Paradebeispiel, weist eine faszinierende Vielfalt zwischen verschiedenen Arten auf. Diese Homologen weisen jedoch spezifische Evolutionsmuster auf, die für die Weiterentwicklung der Proteinstrukturvorhersage von entscheidender Bedeutung sind. Es stellt sich eine zentrale Frage: Welchen Zusammenhang haben diese Modelle mit der Enzymfunktion?
Dr. Xie und Dr. Warshel wollen diesen komplexen Zusammenhang entschlüsseln. Sie betrachten Proteine als eine Sprache aus Aminosäure-„Buchstaben“ und entwickeln generative KI-Tools, die den Prinzipien des weit verbreiteten ChatGPT ähneln.
Ihr KI-Tool quantifiziert die Wahrscheinlichkeit, dass bestimmte Sequenzen oder Mutationen in der Natur vorkommen. Faszinierenderweise fanden sie heraus, dass diese Wahrscheinlichkeit hauptsächlich mit der katalytischen Aktivität des Enzyms zusammenhängt, wenn Mutationen in der Nähe des Substrats auftreten. Umgekehrt korrelieren Mutationen in der Enzymstruktur hauptsächlich mit der Proteinstabilität. Dieses Wissen hat die erfolgreiche Entwicklung mehrerer Proteine, einschließlich Luciferase, ermöglicht.
Dr. Warshel sagte: „Die Beziehung zwischen evolutionären Informationen und verschiedenen Enzymfunktionen legt nahe, dass Enzyme tatsächlich Objekte auf mehreren Ebenen sind.“ Diese Studie hat das Potenzial, unser Verständnis der Enzymkatalyse und -evolution zu revolutionieren.
Diese Forschung wurde jetzt in der Zeitschrift veröffentlicht Nationale wissenschaftliche Zeitschriftbietet nicht nur neue Einblicke in die Enzymkatalyse, sondern verspricht auch praktische Anwendungen in der Biotechnologie und darüber hinaus.
Mehr Informationen:
Wen Jun Xie et al., Nutzung generativer KI zur Entschlüsselung der Enzymkatalyse und -evolution für eine verbesserte Technik, Nationale wissenschaftliche Zeitschrift (2023). DOI: 10.1093/nsr/nwad331
Bereitgestellt von Science China Press
Zitat: Generative KI beleuchtet die Geheimnisse von Enzymen mithilfe von in der Natur entwickelten Sequenzen (20. März 2024), abgerufen am 20. März 2024 von https://phys.org/news/2024-03-generative-ai-illuminates -enzyme-secrets.html
Dieses Dokument unterliegt dem Urheberrecht. Abgesehen von der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.