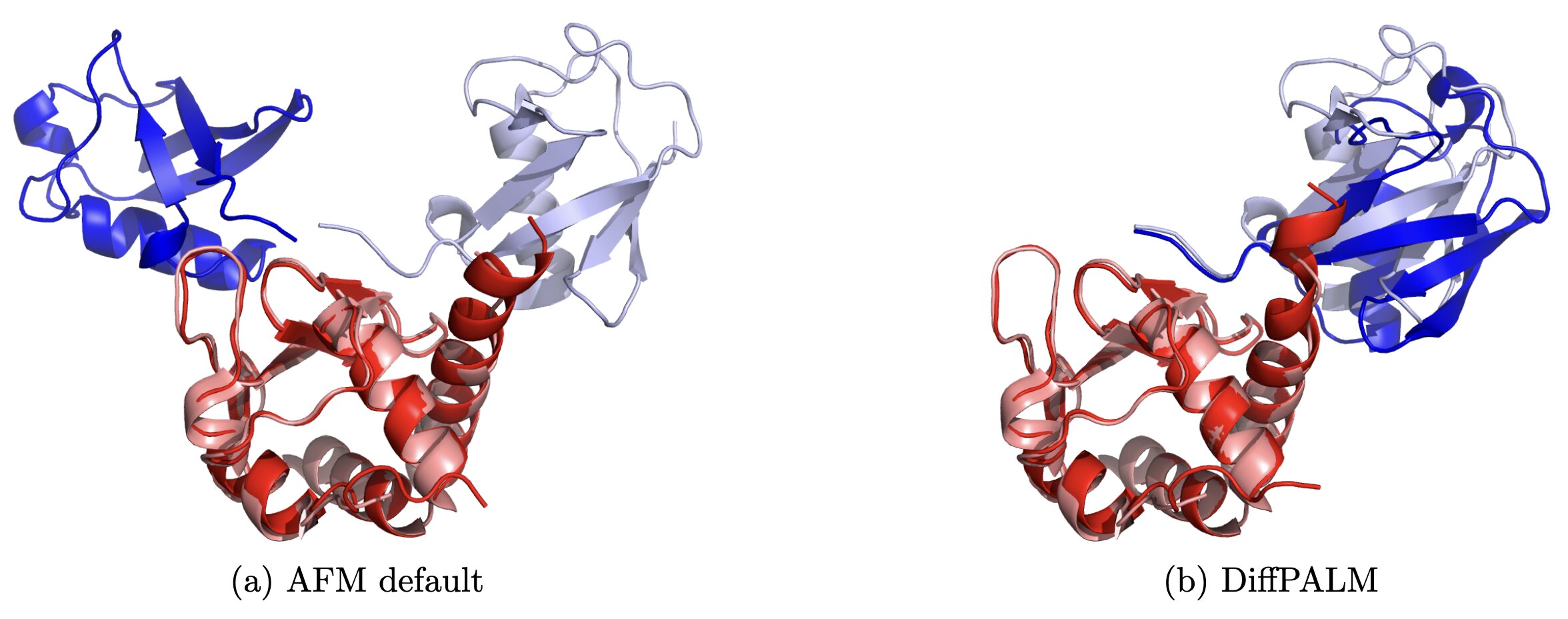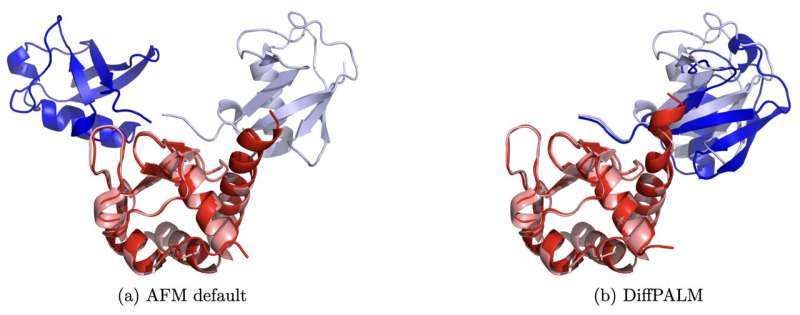
Vergleich der Standard-MSA-Transformer-Matching-Strategie von AFM mit DiffPALM für eine Proteinstruktur. Bildnachweis: Lupo et al. 2024, DOI: 10.1073/pnas.2311887121
Proteine sind die Bausteine des Lebens und an praktisch allen biologischen Prozessen beteiligt. Das Verständnis, wie Proteine miteinander interagieren, ist entscheidend für die Entschlüsselung der Komplexität zellulärer Funktionen und hat erhebliche Auswirkungen auf die Arzneimittelentwicklung und die Behandlung von Krankheiten.
Allerdings ist die Vorhersage, welche Proteine aneinander binden, eine Herausforderung in der Computerbiologie, vor allem aufgrund der großen Vielfalt und Komplexität der Proteinstrukturen. Doch eine neue Studie der Gruppe von Anne-Florence Bitbol an der EPFL könnte die Situation durchaus ändern.
Das Wissenschaftlerteam, darunter Umberto Lupo, Damiano Sgarbossa und Bitbol, entwickelte DiffPALM (Differentiable Pairing using Alignment-based Language Models), einen KI-basierten Ansatz, der die Vorhersage interagierender Proteinsequenzen erheblich vorantreiben kann. Die Studie ist veröffentlicht in PNAS.
DiffPALM nutzt die Leistungsfähigkeit von Protein-Sprachmodellen, einem fortschrittlichen Konzept des maschinellen Lernens, das der Verarbeitung natürlicher Sprache entlehnt ist, um Proteininteraktionen zwischen Mitgliedern zweier Proteinfamilien mit beispielloser Genauigkeit zu analysieren und vorherzusagen.
Es nutzt diese Techniken des maschinellen Lernens, um interagierende Proteinpaare vorherzusagen. Dies führt zu einer deutlichen Verbesserung gegenüber anderen Methoden, die oft große und vielfältige Datensätze erfordern und mit der Komplexität eukaryontischer Proteinkomplexe zu kämpfen haben.
Ein weiterer Vorteil von DiffPALM ist seine Vielseitigkeit, da es auch mit kleineren Sequenzdatensätzen arbeiten und somit mit seltenen Proteinen umgehen kann, die nur wenige Homologe haben, also Proteine aus verschiedenen Arten, die eine gemeinsame evolutionäre Abstammung haben. Es nutzt Protein-Sprachmodelle, die auf Multiple Sequence Alignments (MSA) trainiert wurden, wie das MSA Transformer-Modul von AlphaFold und EvoFormer, die es ihm ermöglichen, komplexe Wechselwirkungen zwischen Proteinen mit hoher Präzision zu verstehen und vorherzusagen.
Darüber hinaus ist der Einsatz von DiffPALM vielversprechend bei der Vorhersage der Struktur von Proteinkomplexen, bei denen es sich um komplexe Strukturen handelt, die durch die Bindung mehrerer Proteine entstehen und für viele zelluläre Prozesse essentiell sind.
In der Studie verglich das Team DiffPALM mit herkömmlichen auf Koevolution basierenden Matching-Methoden, die untersuchen, wie sich Proteinsequenzen im Laufe der Zeit gemeinsam entwickeln, wenn sie eng interagieren: Veränderungen in einem Protein können zu Veränderungen beim Interaktionspartner führen. Dies ist ein äußerst wichtiger Aspekt der Molekular- und Zellbiologie, der durch auf MSAs trainierte Proteinsprachmodelle gut erfasst wird.
Es hat sich gezeigt, dass DiffPALM herkömmliche Methoden bei anspruchsvollen Benchmarks übertrifft und seine Robustheit und Wirksamkeit unter Beweis stellt.
Die Anwendung von DiffPALM ist im Bereich der grundlegenden Proteinbiologie offensichtlich, geht aber darüber hinaus, da es das Potenzial hat, ein leistungsstarkes Werkzeug in der medizinischen Forschung und Arzneimittelentwicklung zu werden. Beispielsweise kann die genaue Vorhersage von Proteininteraktionen dazu beitragen, Krankheitsmechanismen zu verstehen und gezielte Therapien zu entwickeln.
Die Forscher haben DiffPALM frei verfügbar gemacht, in der Hoffnung, dass es von der wissenschaftlichen Gemeinschaft weithin übernommen wird, um die Computerbiologie voranzutreiben und Forschern die Erforschung der Komplexität von Proteininteraktionen zu ermöglichen.
Durch die Kombination fortschrittlicher Techniken des maschinellen Lernens und der effizienten Verwaltung komplexer biologischer Daten stellt DiffPALM einen bedeutenden Fortschritt in der Computerbiologie dar.
Dies verbessert nicht nur unser Verständnis der Proteininteraktionen, sondern eröffnet auch neue Wege in der medizinischen Forschung, die möglicherweise zu Durchbrüchen bei der Behandlung von Krankheiten und der Arzneimittelentwicklung führen.
Mehr Informationen:
Lupo, Umberto et al., Matching interagierender Proteinsequenzen mithilfe maskierter Sprachmodellierung, Verfahren der Nationalen Akademie der Wissenschaften (2024). DOI: 10.1073/pnas.2311887121. doi.org/10.1073/pnas.2311887121
Zur Verfügung gestellt von der Ecole Polytechnique Fédérale de Lausanne
Zitat: KI-basierter Ansatz passt zu Proteininteraktionspartnern (24. Juni 2024), abgerufen am 24. Juni 2024 von https://phys.org/news/2024-06-ai-based-approach-protein-interaction.html
Dieses Dokument unterliegt dem Urheberrecht. Mit Ausnahme der fairen Nutzung für private Studien- oder Forschungszwecke darf kein Teil ohne schriftliche Genehmigung reproduziert werden. Der Inhalt dient lediglich der Information.